# ESEMPI DI USO DI R (per il vol.1 di MaCoSa) [altri usi]
# Le righe non precedute da # possono essere copiate;
# tra queste quelle evidenziate in viola sono d'uso generale.
# Quelle precedute da # sono dei commenti da leggere.
# Quelle in blu (e i grafici) sono esempi di uscite
#
# calcoli e termini ripartizioni finestre e immagini
# funzioni grafici di funzioni di 1 variabile
# grafici per punti altre curve altre statistiche
# calcoli vari figure varie approfondimenti
# Indice alfabetico comandi
#
# COMANDI di base per R
#
# >> CALCOLI E TERMINI <<
# Si possono mettere sigoli termini o più termini separati da ;
5+7; 5-7; 5*7; 5/7; 5^7; 50+7; 50-7; 50*7; 50/7; 50^7; 2+3*5
# si possono assegnare valori a variabili usando <- o ->; quando si
# richiama un termine ne viene riportato il valore (non l'espressione)
a <- 7; 12 -> b; a+b; a*b; pippo <- a*b+a/b+100; pippo
[1] 19
[1] 84
[1] 184.5833
# Se si riassegna una variabile, non vengono automaticamente cambiati
# i valori di varibili che contengano questa, a meno che non venga
# rieffettuata l'assegnazione
a <- 3; b <- 10; ab <- a+b; ab; a <- -4; ab
[1] 13
[1] 13
a <- -4; ab <- a+b; ab
[1] 6
# posso assegnare lo stesso termine assegnato ad una variabile
# assegnando a questa il valore della precedente
x <- 5+7; y <- x; x; y
[1] 12
[1] 12
# Variabili che differiscono per le dimensioni di alcuni caratteri
# sono interpretate come differenti
ab <- 5; AB <- 6; aB <- 7; ab; AB; aB
[1] 5
[1] 6
[1] 7
# Il comando .Last.value riproduce l'ultimo valore calcolato, che
# sia stato o no visualizzato
AA <- 7+10/2
.Last.value+50000
[1] 50012
# Una collezione ordinata di numeri può essere assegnata ad una
# variabile nel modo seguente, e si possono eseguire operazioni su essi
# comandandole sulla variabile; il numero […] indica il numero d'ordine
# della prima uscita che compare sulla riga corrispondente
x <- c(2, 10, 15, 20, 25, 30, 100, 150, 1000, 12345)
x+500+10^6
[1] 1000502 1000510 1000515 1000520 1000525 1000530 1000600 1000650
[9] 1001500 1012845
# Con un ciclo for è possibile assegnare singoli valori alla variabile x;
# man mano possiamo stampare le uscite con print
for (x in c(2,10,15,20,25,30,100,150,1000,12345)) print(x+500+10^6)
[1] 1000502
[1] 1000510
...
[1] 1012845
# In un ciclo for posso variare di 1 in 1 la variabile con un :
for (x in -2:1) print(x)
[1] -2
[1] -1
[1] 0
[1] 1
for (x in (-2:1)*5) print(x)
[1] -10
[1] -5
[1] 0
[1] 5
# Se si scrive library(MASS) si possono usare vari comandi tra cui
# fractions che consente di approssimare numeri decimali con frazioni
120/360; fractions(120/360)
[1] 0.3333333
[1] 1/3
a <- 23; b <- 18; c <- -7; d <- 24
a/b+c/d;fractions(a/b+c/d); c(a*d+c*b,"/",b*d);fractions((a*d+c*b)/(b*d))
[1] 0.9861111
[1] 71/72
[1] "426" "/" "432"
[1] 71/72
# Numeri molto "grandi" o molto "piccoli" vengono scritti in notazione
# esponenziale:
41610000000000
[1] 4.161e+13
1/41610000000000
[1] 2.403268e-14
# un numero scritto viene memorizzato in modo particolare, sia per le
# quantita' di cifre che vengono usate che per il modo in cui
# internamente avviene la registrazione dei numeri; ad esempio dal
# seguente calcolo ci aspetteremmo 0 invece abbiamo:
41610000000000-4.161*10^13
[1] 0.0078125
# invocando la libreria per il calcolo frazionario abbiamo:
library(MASS); fractions(41610000000000-4.161*10^13)
[1] 1/128
# questo valore equivale a 2^-7
2^-7
[1] 0.0078125
# i numeri vengono rappresentati internamente non in base 10 ma in base 2;
# ciò dà luogo a qualche differenza sulle ultime cifre (per 41610000000000
# abbiamo visto la differenza, molto piccola, di 0.0078125)
#
# Schiacciando il tasto freccia ^ [v] si rivedono preced. [segu.] comandi:
# cosa COMODA per rivedere, correggere o modificare precedenti comandi.
#
# >> RIPARTIZIONI <<
# Ecco come rappresentare una ripartizione di dati (vedi LS-1-§3) mediante
# un diagramma a barre (verticale od orizzontale, con eventuali
# rette che indicano le ascisse, tratteggiate con "lty")
dati <- c(116148,9306,35756,45238,58919,168733)
barplot(dati)
barplot(dati,names.arg=c("al","tb","v","ab","tr","a"),ylim=c(0,200e3))
abline(h=c(0,50e3,100e3,150e3,200e3),col="red",lty=3)
barplot(dati,horiz=TRUE)
# i colori usabili sono richiamabili col comando colors()


 # La distribuzione percentuale degli stessi dati
sum(dati); datiperc <- dati/sum(dati)*100; datiperc
etichette <- c("al","tb","v","ab","tr","a")
barplot(datiperc, names.arg=etichette, ylim=c(0,40))
abline(h=c(0,10,20,30,40),col="red",lty=3)
[1] 434100
[1] 26.756047 2.143746 8.236812 10.421101 13.572679 38.869615
# La distribuzione percentuale degli stessi dati
sum(dati); datiperc <- dati/sum(dati)*100; datiperc
etichette <- c("al","tb","v","ab","tr","a")
barplot(datiperc, names.arg=etichette, ylim=c(0,40))
abline(h=c(0,10,20,30,40),col="red",lty=3)
[1] 434100
[1] 26.756047 2.143746 8.236812 10.421101 13.572679 38.869615
 # Ecco una rappresentazione con (vedi LS-1-§4) un diagramma a striscia
barplot(height=cbind(dati/sum(dati)*100),horiz=TRUE)
# Ecco una rappresentazione con (vedi LS-1-§4) un diagramma a striscia
barplot(height=cbind(dati/sum(dati)*100),horiz=TRUE)
 # e con un diagrammi a settori circolari (o a torta - "pie" in inglese)
pie(dati, labels=etichette)
# e con un diagrammi a settori circolari (o a torta - "pie" in inglese)
pie(dati, labels=etichette)
 dati/sum(dati)*360 # ampiezza in gradi dei settori
[1] 96.321769 7.717484 29.652522 37.515964 48.861645 139.930615
# L'areogramma soprastante a destra è stato ottenuto (usando paste,
# ossia "incolla") con:
percentuali <- round(dati/sum(dati)*1000)/10
pie(dati, labels=paste(etichette,":",percentuali,"%"))
# round arrotonda agli interi; quindi round(x*1000)/1000 arrotonda
# ai millesimi: 0.268, 0.021,...,0.389 e round(x*1000)/10 moltiplica
# tali valori per 100: 2.68, 0.21,...,3.89.
#
# >> FINESTRE e IMMAGINI <<
# Se vogliamo dimensionare in modi particolari i grafici, in modo da
# confrontarli, possiamo dimensionare le finestre grafiche col comando
# windows; ad es. windows(3,3) apre una nuova finestra di 3*3 pollici
# in cui viene tracciato il successivo grafico. Questo può essere comodo
# anche per unire e sovrapporre grafici fatti in tempi diversi.
# Ecco come conviene salvare un'immagine. Si clicca su di essa e, dal menu
# che si apre cliccando, la si salva come BitMap e la si incolla su Paint
# (o un altro programma di grafica), si aggiunge quello che si vuole e la si
# ridimensiona. A questo punto o la si copia e salva nel documento in cui si
# sta operando o, da Paint, la si salva in formato GIF, che occupa poco spazio
# e consente di operare successive modifiche con Paint.
#
# >> FUNZIONI <<
# Esempi di funzioni ad 1, 2 e 3 input (vedi LS-2-§3)
perc1 <- function(dato) dato/12450*100
perc2 <- function(dato,totale) dato/totale*100
ripart <- function(dato,totale,base) dato/totale*base
perc1(5400); perc2(5400,12450); ripart(5400,12450,100); ripart(5400,12450,360)
[1] 43.37349
[1] 43.37349
[1] 43.37349
[1] 156.1446
# un altro esempio
k <- 4; f <- function(x) k*x^2; f(3)
[1] 36
# Possiamo comporre più funzioni (sqrt indica la radice quadrata):
F1 <- function(x) x^2; F2 <- function(x) sqrt(x)
F1(F2(5)); F2(F1(5)); F1(F2(-5)); F2(F1(-5))
[1] 5
[1] 5
[1] NaN
Warning message: In sqrt(x) : NaNs produced
[1] 5
#
# >> GRAFICI DI FUNZIONI di 1 variabile <<
# Un modo semplice per tracciare il grafico di una funzione (vedi LS-2-§3),
# usando plot; volendo posso aggiungere abline che (come già visto)
# traccia rette x = h e y = v
F <- function(x) x*360/434100
plot(F, 0,400E3, col="blue")
abline(h=0, col="brown"); abline(v=0, col="brown")
dati/sum(dati)*360 # ampiezza in gradi dei settori
[1] 96.321769 7.717484 29.652522 37.515964 48.861645 139.930615
# L'areogramma soprastante a destra è stato ottenuto (usando paste,
# ossia "incolla") con:
percentuali <- round(dati/sum(dati)*1000)/10
pie(dati, labels=paste(etichette,":",percentuali,"%"))
# round arrotonda agli interi; quindi round(x*1000)/1000 arrotonda
# ai millesimi: 0.268, 0.021,...,0.389 e round(x*1000)/10 moltiplica
# tali valori per 100: 2.68, 0.21,...,3.89.
#
# >> FINESTRE e IMMAGINI <<
# Se vogliamo dimensionare in modi particolari i grafici, in modo da
# confrontarli, possiamo dimensionare le finestre grafiche col comando
# windows; ad es. windows(3,3) apre una nuova finestra di 3*3 pollici
# in cui viene tracciato il successivo grafico. Questo può essere comodo
# anche per unire e sovrapporre grafici fatti in tempi diversi.
# Ecco come conviene salvare un'immagine. Si clicca su di essa e, dal menu
# che si apre cliccando, la si salva come BitMap e la si incolla su Paint
# (o un altro programma di grafica), si aggiunge quello che si vuole e la si
# ridimensiona. A questo punto o la si copia e salva nel documento in cui si
# sta operando o, da Paint, la si salva in formato GIF, che occupa poco spazio
# e consente di operare successive modifiche con Paint.
#
# >> FUNZIONI <<
# Esempi di funzioni ad 1, 2 e 3 input (vedi LS-2-§3)
perc1 <- function(dato) dato/12450*100
perc2 <- function(dato,totale) dato/totale*100
ripart <- function(dato,totale,base) dato/totale*base
perc1(5400); perc2(5400,12450); ripart(5400,12450,100); ripart(5400,12450,360)
[1] 43.37349
[1] 43.37349
[1] 43.37349
[1] 156.1446
# un altro esempio
k <- 4; f <- function(x) k*x^2; f(3)
[1] 36
# Possiamo comporre più funzioni (sqrt indica la radice quadrata):
F1 <- function(x) x^2; F2 <- function(x) sqrt(x)
F1(F2(5)); F2(F1(5)); F1(F2(-5)); F2(F1(-5))
[1] 5
[1] 5
[1] NaN
Warning message: In sqrt(x) : NaNs produced
[1] 5
#
# >> GRAFICI DI FUNZIONI di 1 variabile <<
# Un modo semplice per tracciare il grafico di una funzione (vedi LS-2-§3),
# usando plot; volendo posso aggiungere abline che (come già visto)
# traccia rette x = h e y = v
F <- function(x) x*360/434100
plot(F, 0,400E3, col="blue")
abline(h=0, col="brown"); abline(v=0, col="brown")
 # Un modo per ottenere un grafico migliore (e aggiungerne altri
# sulla stessa scala) è il seguente; si scelgono la parte di piano
# cartesiano [x1,x2]*[y1,y2] assegnandone i valori ad una variabile
# (ad es. di nome view), l'origine (assegnandone le coordinate ad una
# variabile (ad es. orig), e le posizioni delle tacche sugli assi
# attraverso cui far passare la griglia ad altre due (ad es. tx e ty)
view <- c(-10e3,400e3,-10,300); orig <- c(0,0)
tx <- c(0,50,100,150,200,250,300,350,400)*1000
ty <- c(0,50,100,150,200,250,300)
# poi si copiano e incollano i seguenti comandi
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
curve(F, add=TRUE, col="blue", from=0, to=400e3)
# se nell'ultimo comando, "curve", non metto "from" e "to" il grafico
# viene disegnato su tutto l'intervallo scelto per le ascisse
# Un modo per ottenere un grafico migliore (e aggiungerne altri
# sulla stessa scala) è il seguente; si scelgono la parte di piano
# cartesiano [x1,x2]*[y1,y2] assegnandone i valori ad una variabile
# (ad es. di nome view), l'origine (assegnandone le coordinate ad una
# variabile (ad es. orig), e le posizioni delle tacche sugli assi
# attraverso cui far passare la griglia ad altre due (ad es. tx e ty)
view <- c(-10e3,400e3,-10,300); orig <- c(0,0)
tx <- c(0,50,100,150,200,250,300,350,400)*1000
ty <- c(0,50,100,150,200,250,300)
# poi si copiano e incollano i seguenti comandi
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
curve(F, add=TRUE, col="blue", from=0, to=400e3)
# se nell'ultimo comando, "curve", non metto "from" e "to" il grafico
# viene disegnato su tutto l'intervallo scelto per le ascisse
 # Con alcune opzioni è possibile decidere (con "n") il numero di
# punti, decidere se isolarli (con "type"), darne forma ("pch")
# e dimensioni ("cex"). Due esempi:
curve(F,add=TRUE,col="blue",from=0,to=400e3, type="p", n=50)
curve(F,add=TRUE,col="blue",from=0,to=400e3, type="p", n=50, pch=".", cex=3)
# Con alcune opzioni è possibile decidere (con "n") il numero di
# punti, decidere se isolarli (con "type"), darne forma ("pch")
# e dimensioni ("cex"). Due esempi:
curve(F,add=TRUE,col="blue",from=0,to=400e3, type="p", n=50)
curve(F,add=TRUE,col="blue",from=0,to=400e3, type="p", n=50, pch=".", cex=3)
 # Cambiando le scale posso rappresentare altre funzioni:
f <- function(x) 1.45*x; g <- function(x) 4.5*x
view <- c(-1,6,-3,25); orig <- c(0,0)
ty <- c(0,5,10,15,20,25)
tx <- c(-1,0,1,2,3,4,5,6)
# faccio seguire i comandi nelle righe sopra colorate in viola
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
# e poi (add=TRUE serve per poter aggiungere grafici):
curve(g, add=TRUE,col="green4", from=0, to=5)
curve(f, add=TRUE,col="blue", from=0, to=5)
# Cambiando le scale posso rappresentare altre funzioni:
f <- function(x) 1.45*x; g <- function(x) 4.5*x
view <- c(-1,6,-3,25); orig <- c(0,0)
ty <- c(0,5,10,15,20,25)
tx <- c(-1,0,1,2,3,4,5,6)
# faccio seguire i comandi nelle righe sopra colorate in viola
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
# e poi (add=TRUE serve per poter aggiungere grafici):
curve(g, add=TRUE,col="green4", from=0, to=5)
curve(f, add=TRUE,col="blue", from=0, to=5)
 #
# >> GRAFICI per PUNTI <<
# Se voglio rappresentare le seguenti coppie di punti (vedi LS-2-q.18):
# 78, 212
# 79, 226
# 80, 212
# 81, 217
# 82, 283
# metto in x i primi elementi e in y i secondi, scelgo
# la porzione di piano e le tacche:
x <- c(78,79,80,81,82); y <- c(212,226,212,217,283)
view <- c(75,85, -50,300); orig <- c(75,0)
tx <- c(75,76,77,78,79,80,81,82,83,84,85)
ty <- c(-50,0,50,100,150,200,250,300)
# quindi attacco i soliti comandi:
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
# e, infine, uso il comando lines per tracciare una poligonale
# da (78,212) a (79,226) ... a (82,283):
lines(x,y)
#
# >> GRAFICI per PUNTI <<
# Se voglio rappresentare le seguenti coppie di punti (vedi LS-2-q.18):
# 78, 212
# 79, 226
# 80, 212
# 81, 217
# 82, 283
# metto in x i primi elementi e in y i secondi, scelgo
# la porzione di piano e le tacche:
x <- c(78,79,80,81,82); y <- c(212,226,212,217,283)
view <- c(75,85, -50,300); orig <- c(75,0)
tx <- c(75,76,77,78,79,80,81,82,83,84,85)
ty <- c(-50,0,50,100,150,200,250,300)
# quindi attacco i soliti comandi:
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
# e, infine, uso il comando lines per tracciare una poligonale
# da (78,212) a (79,226) ... a (82,283):
lines(x,y)
 # cambiando le righe iniziali ho il grafico sopra a destra:
view <- c(77,83, 200,300); orig <- c(77,200)
tx <- c(77,78,79,80,81,82,83)
ty <- c(200,210,220,230,240,250,260,270,280,290,300)
#
# Un altro esempio (vedi LS-2-e3)
# Mettiamo i punti da rappresentare, assi e tacche:
x <- c(7+55/60, 9+36/60, 9+40/60, 10+41/60, 10+50/60, 12+43/60)
y <- c(0, 219, 219, 316, 316, 626)
view <- c(7,13, -20,650); orig <- c(7,0)
tx <- c(7,8,9,10,11,12,13); ty <- c(0,100,200,300,400,500,600)
# quindi attacchiamo i soliti comandi:
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
# e, infine, oltre a lines, il comando points per evidenziare i singoli
# punti (points traccia punti, senza opzioni mediante dei circoletti,
# altrimenti in svariate forme; con pch="." comandiamo di tracciarli come
# punti tipografici, con cex=4 diamo le dimensioni di essi, con col diamo
# loro il colore)
lines(x,y); points(x,y, pch=".", cex=4, col="blue")
# cambiando le righe iniziali ho il grafico sopra a destra:
view <- c(77,83, 200,300); orig <- c(77,200)
tx <- c(77,78,79,80,81,82,83)
ty <- c(200,210,220,230,240,250,260,270,280,290,300)
#
# Un altro esempio (vedi LS-2-e3)
# Mettiamo i punti da rappresentare, assi e tacche:
x <- c(7+55/60, 9+36/60, 9+40/60, 10+41/60, 10+50/60, 12+43/60)
y <- c(0, 219, 219, 316, 316, 626)
view <- c(7,13, -20,650); orig <- c(7,0)
tx <- c(7,8,9,10,11,12,13); ty <- c(0,100,200,300,400,500,600)
# quindi attacchiamo i soliti comandi:
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
# e, infine, oltre a lines, il comando points per evidenziare i singoli
# punti (points traccia punti, senza opzioni mediante dei circoletti,
# altrimenti in svariate forme; con pch="." comandiamo di tracciarli come
# punti tipografici, con cex=4 diamo le dimensioni di essi, con col diamo
# loro il colore)
lines(x,y); points(x,y, pch=".", cex=4, col="blue")
 #
# Se (vedi LS-2-e20) voglio andare a capo durante la scrittura di un
# comando basta che batta a capo: il comando viene spezzato su più
# righe e viene interpretato correttamente da R
x <- c(0.5,-0.5,-0.5, 1, 1,-1,-1, 1.5, 1.5,-1.5, # continua nella riga successiva
-1.5, 2, 2,-2,-2, 2.5, 2.5)
y <- c(0.5, 0.5,-0.5,-0.5, 1, 1,-1,-1, 1.5, 1.5, # continua
-1.5,-1.5, 2, 2,-2,-2, 2.5)
# Un'alternativa, per caricare molti dati in una variabile, e'
# caricarne una parte e poi aggiungere i rimanenti alla stessa
# variabile con un'altra assegnazione:
x <- c(0.5,-0.5,-0.5, 1, 1,-1,-1, 1.5, 1.5,-1.5)
x <- c(x ,-1.5, 2, 2,-2,-2, 2.5, 2.5)
view <- c(-4,4, -4,4); orig <- c(0,0)
tx <- c(-4,-3,-2,-1,0,1,2,3,4); ty <- tx
# Con asp possiamo dare il rapporto Δy/Δx usato nel grafico;
# con asp=1 otteniamo un sistema monometrico
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="",asp=1)
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
lines(x,y)
#
# Se (vedi LS-2-e20) voglio andare a capo durante la scrittura di un
# comando basta che batta a capo: il comando viene spezzato su più
# righe e viene interpretato correttamente da R
x <- c(0.5,-0.5,-0.5, 1, 1,-1,-1, 1.5, 1.5,-1.5, # continua nella riga successiva
-1.5, 2, 2,-2,-2, 2.5, 2.5)
y <- c(0.5, 0.5,-0.5,-0.5, 1, 1,-1,-1, 1.5, 1.5, # continua
-1.5,-1.5, 2, 2,-2,-2, 2.5)
# Un'alternativa, per caricare molti dati in una variabile, e'
# caricarne una parte e poi aggiungere i rimanenti alla stessa
# variabile con un'altra assegnazione:
x <- c(0.5,-0.5,-0.5, 1, 1,-1,-1, 1.5, 1.5,-1.5)
x <- c(x ,-1.5, 2, 2,-2,-2, 2.5, 2.5)
view <- c(-4,4, -4,4); orig <- c(0,0)
tx <- c(-4,-3,-2,-1,0,1,2,3,4); ty <- tx
# Con asp possiamo dare il rapporto Δy/Δx usato nel grafico;
# con asp=1 otteniamo un sistema monometrico
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="",asp=1)
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
lines(x,y)
 # Vediamo come caricare il file con i dati sul salto in alto (vedi LS-2-§2);
# prima ne esamino le righe iniziali, per controllare che cos'è;
# Uso il comando readLines
readLines("http://macosa.dima.unige.it/om/prg/gfu/altomas.txt", n=5)
[1] "'record alto maschi dal 32 ogni 4 anni" "1932,203"
[3] "1936,207" "1940,209"
[5] "1944,211"
# ok; è una tabella di dati; ogni riga contiene due dati separati
# da una virgola; il comando per leggere la tabella è read.table;
# salto (con skip) la prima riga di commenti e uso "," come separatore;
# vedo le prime tre righe
read.table("http://macosa.dima.unige.it/om/prg/gfu/altomas.txt",sep=",",nrows=3,skip=1)
V1 V2
1 1932 203
2 1936 207
3 1940 209
# se voglio vedere tutto faccio:
read.table("http://macosa.dima.unige.it/om/prg/gfu/altomas.txt",sep=",",skip=1)
V1 V2
1 1932 203
2 1936 207
...
19 2004 245
20 2008 245
# Come vedo, le due variabili vengono chiamate V1 e V2. Metto
# in hm e in hf la tabella dei maschi e quella delle femmine (che
# è in un altro file che potrei esaminare allo stesso modo):
hm <- read.table("http://macosa.dima.unige.it/om/prg/gfu/altomas.txt",sep=",",skip=1)
hf <- read.table("http://macosa.dima.unige.it/om/prg/gfu/altofem.txt",sep=",",skip=1)
# il grafico per punti dei primi dati lo posso ottenre così (hm$V1 contiene
# i valori di V1 della tabella hm; hm$V2 contiene ...)
plot(hm$V1,hm$V2)
# Vediamo come caricare il file con i dati sul salto in alto (vedi LS-2-§2);
# prima ne esamino le righe iniziali, per controllare che cos'è;
# Uso il comando readLines
readLines("http://macosa.dima.unige.it/om/prg/gfu/altomas.txt", n=5)
[1] "'record alto maschi dal 32 ogni 4 anni" "1932,203"
[3] "1936,207" "1940,209"
[5] "1944,211"
# ok; è una tabella di dati; ogni riga contiene due dati separati
# da una virgola; il comando per leggere la tabella è read.table;
# salto (con skip) la prima riga di commenti e uso "," come separatore;
# vedo le prime tre righe
read.table("http://macosa.dima.unige.it/om/prg/gfu/altomas.txt",sep=",",nrows=3,skip=1)
V1 V2
1 1932 203
2 1936 207
3 1940 209
# se voglio vedere tutto faccio:
read.table("http://macosa.dima.unige.it/om/prg/gfu/altomas.txt",sep=",",skip=1)
V1 V2
1 1932 203
2 1936 207
...
19 2004 245
20 2008 245
# Come vedo, le due variabili vengono chiamate V1 e V2. Metto
# in hm e in hf la tabella dei maschi e quella delle femmine (che
# è in un altro file che potrei esaminare allo stesso modo):
hm <- read.table("http://macosa.dima.unige.it/om/prg/gfu/altomas.txt",sep=",",skip=1)
hf <- read.table("http://macosa.dima.unige.it/om/prg/gfu/altofem.txt",sep=",",skip=1)
# il grafico per punti dei primi dati lo posso ottenre così (hm$V1 contiene
# i valori di V1 della tabella hm; hm$V2 contiene ...)
plot(hm$V1,hm$V2)
 # per rappresentare meglio i dati procedo nel solito modo:
tx <- 1900+c(3,4,5,6,7,8,9,10,11)*10
ty <- 160+c(0,1,2,3,4,5,6,7,8,9,10)*10
view <- c(1930,2010, 160,250); orig <- c(1930,160)
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
lines(hm$V1,hm$V2); lines(hf$V1,hf$V2)
points(hm$V1,hm$V2, pch=".", cex=4, col="blue")
points(hf$V1,hf$V2, pch=".", cex=4, col="red")
# per rappresentare meglio i dati procedo nel solito modo:
tx <- 1900+c(3,4,5,6,7,8,9,10,11)*10
ty <- 160+c(0,1,2,3,4,5,6,7,8,9,10)*10
view <- c(1930,2010, 160,250); orig <- c(1930,160)
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
lines(hm$V1,hm$V2); lines(hf$V1,hf$V2)
points(hm$V1,hm$V2, pch=".", cex=4, col="blue")
points(hf$V1,hf$V2, pch=".", cex=4, col="red")
 # Per rappresentare i numeri indici ne metto i valori in indm e
indf e poi procedo nel solito modo, cambiando scala e tacche:
indm <- hm$V2/hm$V2[1]*100; indf <- hf$V2/hf$V2[1]*100
ty <- 100+c(0,5,10,15,20,25,30)
view <- c(1930,2010, 100,130); orig <- c(1930,100)
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
lines(hm$V1,indm); lines(hf$V1,indf)
points(hm$V1,indm, pch=".", cex=4, col="blue")
points(hf$V1,indf, pch=".", cex=4, col="red")
# Per rappresentare i numeri indici ne metto i valori in indm e
indf e poi procedo nel solito modo, cambiando scala e tacche:
indm <- hm$V2/hm$V2[1]*100; indf <- hf$V2/hf$V2[1]*100
ty <- 100+c(0,5,10,15,20,25,30)
view <- c(1930,2010, 100,130); orig <- c(1930,100)
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
lines(hm$V1,indm); lines(hf$V1,indf)
points(hm$V1,indm, pch=".", cex=4, col="blue")
points(hf$V1,indf, pch=".", cex=4, col="red")
 #
# >> ALTRE CURVE <<
# Vediamo, prima, come fare il grafico di più funzioni (vedi LA-2-q.36):
# basta aggiungere più comandi plot.
F <- function(x) x*1.25; G <- function(x) x*0.8
view <- c(-25,160, -25,160); orig <- c(0,0)
tx <- c(-2,-1,0,1,2,3,4,5,6,7,8,9,10,11,12,13,14,15,16)*10; ty <- tx
# i soliti comandi:
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
# quindi:
curve(F, add=TRUE,col="blue"); curve(G, add=TRUE,col="red")
H <- function(x) F(G(x)); K <- function(x) G(F(x))
curve(H, add=TRUE,col="green"); curve(K, add=TRUE,col="magenta")
#
# >> ALTRE CURVE <<
# Vediamo, prima, come fare il grafico di più funzioni (vedi LA-2-q.36):
# basta aggiungere più comandi plot.
F <- function(x) x*1.25; G <- function(x) x*0.8
view <- c(-25,160, -25,160); orig <- c(0,0)
tx <- c(-2,-1,0,1,2,3,4,5,6,7,8,9,10,11,12,13,14,15,16)*10; ty <- tx
# i soliti comandi:
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
# quindi:
curve(F, add=TRUE,col="blue"); curve(G, add=TRUE,col="red")
H <- function(x) F(G(x)); K <- function(x) G(F(x))
curve(H, add=TRUE,col="green"); curve(K, add=TRUE,col="magenta")
 #
# Per fare il grafico di una curva che non sia una funzione (vedi LA-2-e8)
# posso usare il comando lines. Ecco il grafico della relazione
# inversa di una funzione (spezzo il grafico in 1000 segmentini)
view <- c(-4,10, -4,10); orig <- c(0,0)
tx <- c(-4,-3,-2,-1,0,1,2,3,4,5,6,7,8,9,10); ty <- tx
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="",asp=1)
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
f <- function(x) x^2; curve(f,add=TRUE,col="blue")
t1<-view[1]; t2<-view[2]; punti<-1000; t<- seq(t1,t2,(t2-t1)/punti)
lines(f(t),t,col="green4")
#
# Per fare il grafico di una curva che non sia una funzione (vedi LA-2-e8)
# posso usare il comando lines. Ecco il grafico della relazione
# inversa di una funzione (spezzo il grafico in 1000 segmentini)
view <- c(-4,10, -4,10); orig <- c(0,0)
tx <- c(-4,-3,-2,-1,0,1,2,3,4,5,6,7,8,9,10); ty <- tx
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="",asp=1)
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
f <- function(x) x^2; curve(f,add=TRUE,col="blue")
t1<-view[1]; t2<-view[2]; punti<-1000; t<- seq(t1,t2,(t2-t1)/punti)
lines(f(t),t,col="green4")
 #
# >> PRIME STATISTICHE <<
# Elaborazioni riferite a LS-3-e9,…; è evidente
# che cosa fanno i vari comandi.
alu <- c(156,168,162,150,167,157,170,157,159,164,157,165,
163,165,166,160,163,162,155)
length(alu);sum(alu); min(alu);max(alu); range(alu);sort(alu); mean(alu);median(alu)
[1] 19
[1] 3066
[1] 150
[1] 170
[1] 150 170
[1] 150 155 156 157 157 157 159 160 162 162 163 163 164 165 165 166 167
[18] 168 170
[1] 161.3684
[1] 162
# Il comando che segue rappresenta automaticamente i dati su una specie
# di istogramma, riportando anche i valori dei dati ("stem" è il gambo,
# su cui poggiano le foglie)
stem(alu)
The decimal point is 1 digit(s) to the right of the |
15 | 0
15 | 567779
16 | 022334
16 | 55678
17 | 0
# summary riporta min e max, media e mediana, 1° e 3° quartile, ossia
# il 25° e il 75° percentile
summary(alu)
Min. 1st Qu. Median Mean 3rd Qu. Max.
150.0 157.0 162.0 161.4 165.0 170.0
# A differenza di barplot, hist traccia l'istogramma di dati che, qui,
# vengono classificati mediante seq (che abbiamo già usato): seq(A,B,h)
# genera la sequenza di dati che va, di h in h, da A a B; l'opzione
# right=FALSE specifica che i sottintervalli sono del tipo [.,.), ossia
# aperti a destra (e chiusi a sinistra)
# boxplot traccia il box-plot (con l'opzione scelta, orizzontale)
hist(alu,seq(150,171,3),right=FALSE,col="yellow",xlab="",ylab="",main="")
boxplot(alu, horizontal=TRUE)
#
# >> PRIME STATISTICHE <<
# Elaborazioni riferite a LS-3-e9,…; è evidente
# che cosa fanno i vari comandi.
alu <- c(156,168,162,150,167,157,170,157,159,164,157,165,
163,165,166,160,163,162,155)
length(alu);sum(alu); min(alu);max(alu); range(alu);sort(alu); mean(alu);median(alu)
[1] 19
[1] 3066
[1] 150
[1] 170
[1] 150 170
[1] 150 155 156 157 157 157 159 160 162 162 163 163 164 165 165 166 167
[18] 168 170
[1] 161.3684
[1] 162
# Il comando che segue rappresenta automaticamente i dati su una specie
# di istogramma, riportando anche i valori dei dati ("stem" è il gambo,
# su cui poggiano le foglie)
stem(alu)
The decimal point is 1 digit(s) to the right of the |
15 | 0
15 | 567779
16 | 022334
16 | 55678
17 | 0
# summary riporta min e max, media e mediana, 1° e 3° quartile, ossia
# il 25° e il 75° percentile
summary(alu)
Min. 1st Qu. Median Mean 3rd Qu. Max.
150.0 157.0 162.0 161.4 165.0 170.0
# A differenza di barplot, hist traccia l'istogramma di dati che, qui,
# vengono classificati mediante seq (che abbiamo già usato): seq(A,B,h)
# genera la sequenza di dati che va, di h in h, da A a B; l'opzione
# right=FALSE specifica che i sottintervalli sono del tipo [.,.), ossia
# aperti a destra (e chiusi a sinistra)
# boxplot traccia il box-plot (con l'opzione scelta, orizzontale)
hist(alu,seq(150,171,3),right=FALSE,col="yellow",xlab="",ylab="",main="")
boxplot(alu, horizontal=TRUE)
 # Vediamo come confrontare più boxplot
alu2 <- c(172,162,150,167,157,170,157,159,164,157,165,163,165,
166,160,163,162,155)
alu3 <- c(172,162,160,167,157,170,157,159,164,157,165,163,165,
166,160,163,162,180)
boxplot(alu, alu2, alu3)
# a destra la figura ottenuta con:
boxplot(alu, alu2, alu3, col="grey80",show.names=FALSE)
# Vediamo come confrontare più boxplot
alu2 <- c(172,162,150,167,157,170,157,159,164,157,165,163,165,
166,160,163,162,155)
alu3 <- c(172,162,160,167,157,170,157,159,164,157,165,163,165,
166,160,163,162,180)
boxplot(alu, alu2, alu3)
# a destra la figura ottenuta con:
boxplot(alu, alu2, alu3, col="grey80",show.names=FALSE)
 # Volendo possiamo anche sovrapporre più istogrammi, differenziandone
# l'aspetto con vari comandi; ecco un esempio:
interv <- seq(150,183,3); tacchey <- seq(0,6,1)
hist(alu,interv,right=FALSE,col="yellow",xlab="",ylab="",main="",axes=FALSE)
axis(1,pos=0,col="blue",label=TRUE, at=interv)
axis(2,pos=150,col="blue",label=TRUE, at=tacchey)
hist(alu2,interv,right=FALSE,xlab="",ylab="",main="",border="blue",add=TRUE,angle=135,density=14)
hist(alu3,interv,right=FALSE,xlab="",ylab="",main="",border="red",add=TRUE,angle=45,density=7)
# add=TRUE consente di aggiungere un istogramma alla figura già tracciata;
# angle e density fanno tracciare segmenti sulle colonne con varia
# inclinazione (in gradi) e varia densità (segmenti per pollice)
# axes=FALSE mi consente di non tracciare gli assi automaticamente;
# li ho tracciati col comando axis
# Volendo possiamo anche sovrapporre più istogrammi, differenziandone
# l'aspetto con vari comandi; ecco un esempio:
interv <- seq(150,183,3); tacchey <- seq(0,6,1)
hist(alu,interv,right=FALSE,col="yellow",xlab="",ylab="",main="",axes=FALSE)
axis(1,pos=0,col="blue",label=TRUE, at=interv)
axis(2,pos=150,col="blue",label=TRUE, at=tacchey)
hist(alu2,interv,right=FALSE,xlab="",ylab="",main="",border="blue",add=TRUE,angle=135,density=14)
hist(alu3,interv,right=FALSE,xlab="",ylab="",main="",border="red",add=TRUE,angle=45,density=7)
# add=TRUE consente di aggiungere un istogramma alla figura già tracciata;
# angle e density fanno tracciare segmenti sulle colonne con varia
# inclinazione (in gradi) e varia densità (segmenti per pollice)
# axes=FALSE mi consente di non tracciare gli assi automaticamente;
# li ho tracciati col comando axis
 # L'esercizio LS-3-e11 fa riferimento ad un file; prima di caricarlo conviene
# esaminarlo; col comando readLines possiamo leggerne un po' di righe,
# ad es. 5, per vedere come è fatto
readLines("http://macosa.dima.unige.it/om/prg/stf/t-sec.stf", n=5)
[1] "'commento: misure delle durata di 1 sec cronometrate manualmente"
[2] "111"
[3] "103"
[4] "109"
[5] "97"
# Con scan posso, volendo, esaminarlo (in questo caso i dati sono
# pochi e possiamo farlo), e posso caricarlo in una variabile, ad es.
# DATI; in ogni caso salto (con skip=1) la prima riga, di commento
DATI <- scan("http://macosa.dima.unige.it/om/prg/stf/t-sec.stf", skip=1)
length(DATI)
[1] 47
DATI
[1] 111 103 109 97 99 110 99 103 109 106 109 112 98 110
[15] 90 98 96 90 96 90 109 103 96 97 96 96 109 103
[29] 98 94 115 78 85 96 90 121 98 103 97 90 98 129
[43] 96 68 91 80 102
# I dati era tempi in secondi, troncati agli interi. Per avere
# rappresentazioni corrette possiamo aggiungere 1/2 ad essi:
DATI <- DATI+1/2; DATI
[1] 111.5 103.5 109.5 97.5 99.5 110.5 99.5 103.5 109.5
[10] 106.5 109.5 112.5 98.5 110.5 90.5 98.5 96.5 90.5
[19] 96.5 90.5 109.5 103.5 96.5 97.5 96.5 96.5 109.5
[28] 103.5 98.5 94.5 115.5 78.5 85.5 96.5 90.5 121.5
[37] 98.5 103.5 97.5 90.5 98.5 129.5 96.5 68.5 91.5
[46] 80.5 102.5
min(DATI); max(DATI); mean(DATI); median(DATI)
[1] 68.5
[1] 129.5
[1] 99.92553
[1] 98.5
# l'istogramma, in classi ampie 10, e il box-plot, in giallo.
hist(DATI,seq(65,135,10),right=FALSE,col="yellow",xlab="",main="")
abline(h=c(5,10,15,20,25),col="red",lty=3)
# la griglia è stata aggiunta con i soliti comandi.
boxplot(DATI, horizontal=TRUE, col="yellow")
# L'esercizio LS-3-e11 fa riferimento ad un file; prima di caricarlo conviene
# esaminarlo; col comando readLines possiamo leggerne un po' di righe,
# ad es. 5, per vedere come è fatto
readLines("http://macosa.dima.unige.it/om/prg/stf/t-sec.stf", n=5)
[1] "'commento: misure delle durata di 1 sec cronometrate manualmente"
[2] "111"
[3] "103"
[4] "109"
[5] "97"
# Con scan posso, volendo, esaminarlo (in questo caso i dati sono
# pochi e possiamo farlo), e posso caricarlo in una variabile, ad es.
# DATI; in ogni caso salto (con skip=1) la prima riga, di commento
DATI <- scan("http://macosa.dima.unige.it/om/prg/stf/t-sec.stf", skip=1)
length(DATI)
[1] 47
DATI
[1] 111 103 109 97 99 110 99 103 109 106 109 112 98 110
[15] 90 98 96 90 96 90 109 103 96 97 96 96 109 103
[29] 98 94 115 78 85 96 90 121 98 103 97 90 98 129
[43] 96 68 91 80 102
# I dati era tempi in secondi, troncati agli interi. Per avere
# rappresentazioni corrette possiamo aggiungere 1/2 ad essi:
DATI <- DATI+1/2; DATI
[1] 111.5 103.5 109.5 97.5 99.5 110.5 99.5 103.5 109.5
[10] 106.5 109.5 112.5 98.5 110.5 90.5 98.5 96.5 90.5
[19] 96.5 90.5 109.5 103.5 96.5 97.5 96.5 96.5 109.5
[28] 103.5 98.5 94.5 115.5 78.5 85.5 96.5 90.5 121.5
[37] 98.5 103.5 97.5 90.5 98.5 129.5 96.5 68.5 91.5
[46] 80.5 102.5
min(DATI); max(DATI); mean(DATI); median(DATI)
[1] 68.5
[1] 129.5
[1] 99.92553
[1] 98.5
# l'istogramma, in classi ampie 10, e il box-plot, in giallo.
hist(DATI,seq(65,135,10),right=FALSE,col="yellow",xlab="",main="")
abline(h=c(5,10,15,20,25),col="red",lty=3)
# la griglia è stata aggiunta con i soliti comandi.
boxplot(DATI, horizontal=TRUE, col="yellow")
 #
# Sotto, il box-plot che avrei ottenuto con Stat:
··········|------------|=|========|----|············
+-----------------------------+------------------------+
65 100 130
# la sintesi, e il 5° e il 95° percentile
summary(DATI); quantile(DATI,0.05); quantile(DATI,0.95)
Min. 1st Qu. Median Mean 3rd Qu. Max.
68.50 96.50 98.50 99.93 108.00 129.50
5% 82
95% 114.6
# NOTA: Stat traccia i boxplot segnando pure il 5º e il 95º percentile;
# R traccia solo il 25º e il 75º percentile evidenziando poi con dei
# pallini gli eventuali dati che precedono il 25º o seguono il 75º per
# più di 1.5 volte la distanza interquartile, dando in modo alternativo
# l'idea della eventuale dispersione dei dati. Volendo, con l'opzione
# RANGE=0 si elimina questa visualizzazione; vedi il secondo boxplot:
#
# Sotto, il box-plot che avrei ottenuto con Stat:
··········|------------|=|========|----|············
+-----------------------------+------------------------+
65 100 130
# la sintesi, e il 5° e il 95° percentile
summary(DATI); quantile(DATI,0.05); quantile(DATI,0.95)
Min. 1st Qu. Median Mean 3rd Qu. Max.
68.50 96.50 98.50 99.93 108.00 129.50
5% 82
95% 114.6
# NOTA: Stat traccia i boxplot segnando pure il 5º e il 95º percentile;
# R traccia solo il 25º e il 75º percentile evidenziando poi con dei
# pallini gli eventuali dati che precedono il 25º o seguono il 75º per
# più di 1.5 volte la distanza interquartile, dando in modo alternativo
# l'idea della eventuale dispersione dei dati. Volendo, con l'opzione
# RANGE=0 si elimina questa visualizzazione; vedi il secondo boxplot:
 # L'analisi con R dei dati di LS-3-e12:
readLines("http://macosa.dima.unige.it/om/prg/stf/t-arrivi.stf", n=5)
[1] "'commento: distanze temporali tra arrivi successivi a uno sportello"
[2] "15"
[3] "29"
[4] "4"
[5] "99"
DATI <- scan("http://macosa.dima.unige.it/om/prg/stf/t-arrivi.stf", skip=1)
length(DATI)
[1] 134
DATI <- DATI+1/2
min(DATI); max(DATI); mean(DATI); median(DATI)
[1] 1.5
[1] 173.5
[1] 29.55224
[1] 21.5
hist(DATI,seq(0,180,10),right=FALSE,col="yellow",xlab="",main="")
# L'analisi con R dei dati di LS-3-e12:
readLines("http://macosa.dima.unige.it/om/prg/stf/t-arrivi.stf", n=5)
[1] "'commento: distanze temporali tra arrivi successivi a uno sportello"
[2] "15"
[3] "29"
[4] "4"
[5] "99"
DATI <- scan("http://macosa.dima.unige.it/om/prg/stf/t-arrivi.stf", skip=1)
length(DATI)
[1] 134
DATI <- DATI+1/2
min(DATI); max(DATI); mean(DATI); median(DATI)
[1] 1.5
[1] 173.5
[1] 29.55224
[1] 21.5
hist(DATI,seq(0,180,10),right=FALSE,col="yellow",xlab="",main="")
 # per avere l'istogramma delle densità di frequenza:
> hist(DATI,seq(0,180,10),right=FALSE,col="yellow",xlab="",main="",probability=TRUE)
# che poi rappresento scegliendo la scala verticale
hist(DATI,seq(0,180,10),ylim=c(0,0.03),right=FALSE,col="yellow",xlab="",main="",probability=TRUE)
# per avere l'istogramma delle densità di frequenza:
> hist(DATI,seq(0,180,10),right=FALSE,col="yellow",xlab="",main="",probability=TRUE)
# che poi rappresento scegliendo la scala verticale
hist(DATI,seq(0,180,10),ylim=c(0,0.03),right=FALSE,col="yellow",xlab="",main="",probability=TRUE)
 # Sull'istogramma è sovrapposto il grafico dell'esponenziale negativa
# (discussa nella guida alla scheda) e la posizione di media e mediana, cose
# ottenute coi comandi seguenti:
w <- 1/mean(DATI); f <- function(x) w*exp(-w*x)
plot(f,0,180,add=TRUE)
abline(v=mean(DATI),col="red"); abline(v=median(DATI),col="blue")
#
# Ecco come analizzare con R i dati di LS-3-e13, già classificati in
# intervalli
# Metto in una variabile (freq) le frequenze, in un'altra (num_int)
# il numero di intervalli, in una terza (interv) gli estremi degli
# intervallo, e poi metto la riga successiva (le righe in viola le
# copio e incollo, senza preoccuparmi di capirne i dettagli)
morti <- c(46,25,58,186,870,1071,3124)
interv <- c(0,1,15,25,45,65,75,100) # un dato in piu' di freq
totale <- sum(morti); fr_perc <- morti/totale*100; fr_perc
[1] 0.8550186 0.4646840 1.0780669 3.4572491 16.1710037 19.9070632
[7] 58.0669145
# Arrotondo: metto digits=2 in quanto il dato più piccolo 0.464... lo
# voglio arrotondare a 0.46, ossia a 2 cifre
print(fr_perc,digits=2)
[1] 0.86 0.46 1.08 3.46 16.17 19.91 58.07
# porto i dati a 500 mila per migliorare l'elaborazione
rap <- 1e5/sum(morti); freq <- morti*rap; num_int <- length(freq)
dati <- c(seq(interv[1],interv[2],by=(interv[2]-interv[1])/freq[1]))
for(i in 2:num_int){dati<- c(dati,seq(interv[i],interv[i+1],by=(interv[i+1]-interv[i])/freq[i]))}
hist(dati,interv,right=FALSE,xlab="",main="")
# Sull'istogramma è sovrapposto il grafico dell'esponenziale negativa
# (discussa nella guida alla scheda) e la posizione di media e mediana, cose
# ottenute coi comandi seguenti:
w <- 1/mean(DATI); f <- function(x) w*exp(-w*x)
plot(f,0,180,add=TRUE)
abline(v=mean(DATI),col="red"); abline(v=median(DATI),col="blue")
#
# Ecco come analizzare con R i dati di LS-3-e13, già classificati in
# intervalli
# Metto in una variabile (freq) le frequenze, in un'altra (num_int)
# il numero di intervalli, in una terza (interv) gli estremi degli
# intervallo, e poi metto la riga successiva (le righe in viola le
# copio e incollo, senza preoccuparmi di capirne i dettagli)
morti <- c(46,25,58,186,870,1071,3124)
interv <- c(0,1,15,25,45,65,75,100) # un dato in piu' di freq
totale <- sum(morti); fr_perc <- morti/totale*100; fr_perc
[1] 0.8550186 0.4646840 1.0780669 3.4572491 16.1710037 19.9070632
[7] 58.0669145
# Arrotondo: metto digits=2 in quanto il dato più piccolo 0.464... lo
# voglio arrotondare a 0.46, ossia a 2 cifre
print(fr_perc,digits=2)
[1] 0.86 0.46 1.08 3.46 16.17 19.91 58.07
# porto i dati a 500 mila per migliorare l'elaborazione
rap <- 1e5/sum(morti); freq <- morti*rap; num_int <- length(freq)
dati <- c(seq(interv[1],interv[2],by=(interv[2]-interv[1])/freq[1]))
for(i in 2:num_int){dati<- c(dati,seq(interv[i],interv[i+1],by=(interv[i+1]-interv[i])/freq[i]))}
hist(dati,interv,right=FALSE,xlab="",main="")
 # In realtà l'istogramma è stato colorato:
hist(dati,interv,right=FALSE,col="yellow",xlab="",main="")
# Ecco come aggiungere una "griglia"
abline(v=interv,h=seq(0.005,0.03,0.005),lty=3,col="grey50")
# In realtà l'istogramma è stato colorato:
hist(dati,interv,right=FALSE,col="yellow",xlab="",main="")
# Ecco come aggiungere una "griglia"
abline(v=interv,h=seq(0.005,0.03,0.005),lty=3,col="grey50")
 mean(dati); median(dati)
# Alternativa:
tacchey <- c(0,0.005,0.01,0.015,0.02,0.025)
hist(dati,interv,col="yellow",axes=FALSE,xlab="",main="",ylim=c(0,max(tacchey)))
abline(h = tacchey, col="grey60",lty=3)
axis(1,pos=0,col="blue",label=TRUE, at=interv)
axis(2,pos=interv[1],col="blue",label=TRUE, at=tacchey)
mean(dati); median(dati)
# Alternativa:
tacchey <- c(0,0.005,0.01,0.015,0.02,0.025)
hist(dati,interv,col="yellow",axes=FALSE,xlab="",main="",ylim=c(0,max(tacchey)))
abline(h = tacchey, col="grey60",lty=3)
axis(1,pos=0,col="blue",label=TRUE, at=interv)
axis(2,pos=interv[1],col="blue",label=TRUE, at=tacchey)
 # modifico i dati (e copio le altre righe); ottengo:
morti <- c(58,186,870,1071,3124); interv <- c(15,25,45,65,75,100)
...
# modifico i dati (e copio le altre righe); ottengo:
morti <- c(58,186,870,1071,3124); interv <- c(15,25,45,65,75,100)
...
 #
# altezze dei ventenni italiani nel 1976, discusse in vedi LS-3-§4
# analisi dei dati, svolta come sopra
num_int <- 9; freq <- c(7,29,112,240,289,204,89,25,5)
interv <- c(150,155,160,165,170,175,180,185,190,200)
totale <- sum(freq); fr_perc <- freq/totale*100; fr_perc
[1] 0.7 2.9 11.2 24.0 28.9 20.4 8.9 2.5 0.5
dati <- c(seq(interv[1],interv[2],by=(interv[2]-interv[1])/freq[1]))
for (i in 2:num_int) {dati <- c(dati,seq(interv[i],interv[i+1],by=(interv[i+1]-interv[i])/freq[i]))}
hist(dati,interv)
# ottenuto l'istogramma sotto a sinistra, posso scegliere la
# scala verticale e proseguire ottenedo l'istogramma destro
#
# altezze dei ventenni italiani nel 1976, discusse in vedi LS-3-§4
# analisi dei dati, svolta come sopra
num_int <- 9; freq <- c(7,29,112,240,289,204,89,25,5)
interv <- c(150,155,160,165,170,175,180,185,190,200)
totale <- sum(freq); fr_perc <- freq/totale*100; fr_perc
[1] 0.7 2.9 11.2 24.0 28.9 20.4 8.9 2.5 0.5
dati <- c(seq(interv[1],interv[2],by=(interv[2]-interv[1])/freq[1]))
for (i in 2:num_int) {dati <- c(dati,seq(interv[i],interv[i+1],by=(interv[i+1]-interv[i])/freq[i]))}
hist(dati,interv)
# ottenuto l'istogramma sotto a sinistra, posso scegliere la
# scala verticale e proseguire ottenedo l'istogramma destro
 tacchey <- c(0,0.5,1,1.5,2,2.5,3,3.5,4,4.5,5,5.5,6)/100
hist(dati,interv,col="yellow",axes=FALSE,xlab="",main="",ylim=c(0,max(tacchey)))
abline(h = tacchey, col="grey60",lty=3)
axis(1,pos=0,col="blue",label=TRUE, at=interv)
axis(2,pos=interv[1],col="blue",label=TRUE, at=tacchey)
# altre valutazioni statistiche dei dati
summary(dati)
Min. 1st Qu. Median Mean 3rd Qu. Max.
150.0 167.1 171.9 172.0 176.8 200.0
media: 172.0025 mediana = 171.9377163
5% :160.625 25% :167.125 50% :171.9377163
95% :183.8764045 75% :176.7892157
···········|------|====|====|------|·················
tacchey <- c(0,0.5,1,1.5,2,2.5,3,3.5,4,4.5,5,5.5,6)/100
hist(dati,interv,col="yellow",axes=FALSE,xlab="",main="",ylim=c(0,max(tacchey)))
abline(h = tacchey, col="grey60",lty=3)
axis(1,pos=0,col="blue",label=TRUE, at=interv)
axis(2,pos=interv[1],col="blue",label=TRUE, at=tacchey)
# altre valutazioni statistiche dei dati
summary(dati)
Min. 1st Qu. Median Mean 3rd Qu. Max.
150.0 167.1 171.9 172.0 176.8 200.0
media: 172.0025 mediana = 171.9377163
5% :160.625 25% :167.125 50% :171.9377163
95% :183.8764045 75% :176.7892157
···········|------|====|====|------|·················
 #
# >> CALCOLI VARI <<
# Struttura di un termine (vedi LA-3-§2)
G <- function(x) (2^3*-x)/(9-1)+x*5; G(3); G(-5)
[1] 12
[1] -20
x <- 3; v0<-2^3; v1<- -x; v2<-v0*v1; v3<-9-1; v4<-v2/v3; v5<-x*5; v6<-v4+v5; v6
[1] 12
x <- -5; v0<-2^3; v1<- -x; v2<-v0*v1; v3<-9-1; v4<-v2/v3; v5<-x*5; v6<-v4+v5; v6
[1] -20
# Divisori e scomposizione in numeri primi (vedi LA-3-§4); floor(x) dà
# il massimo intero minore o uguale ad x; in una condizione, come quella
# che segue un if, occorre usare == invece di =.
n <- 1580; for(k in 1:n) if (n/k==floor(n/k)) print(k)
[1] 1
[1] 2
[1] 4
[1] 5
[1] 10
[1] 20
[1] 79
[1] 158
[1] 316
[1] 395
[1] 790
[1] 1580
# Occorre usare { e }quando si vuole delimitare l'esecuzione ad alcuni
# comandi; nell'esempio che segue servono per far eseguire print(k)
# e n<-n/k solo se la condizione è verificata
# while significa "fintanto che"
n<-1580; k<-2; while (k<=n) if (n/k==floor(n/k)) {print(k); n<-n/k} else k<-k+1
[1] 2
[1] 2
[1] 5
[1] 79
# La distanza dall'origine (vedi LA-3-e8) è definibile:
D <- function(x,y) sqrt(x^2+y^2)
D(1,2)
[1] 2.236068
# viene visualizzata un'approssimazione del risultato; il programma
# ne elabora in realtà una più precisa (2.236067977...), come si
# vede trasformando l'uscita in forma intera:
for (i in 8:12) print(D(1,2)*10^i)
[1] 223606798
[1] 2236067977
[1] 22360679775
[1] 223606797750
[1] 2.236068e+12
# In R le assegnazioni (vedi AL-§2) sono fatte usando <- o ->;
# occorre sempre esplicitare il simbolo di moltiplicazione, usando *
2·2
Error: unexpected input in "2·"
> 2*2
[1] 4
# Come già visto, chiamando la libreria MASS è possibile approssimare
# i numeri decimali mediante frazioni
library(MASS)
fractions(0.140140140140140140140); fractions(10.45454545454545454545)
[1] 140/999
[1] 115/11
# per passare da una forma frazionaria generata da fractions ad
# un numero usuale si può usare as.numeric:
x <- fractions(10.45454545454545454545); x
[1] 115/11
y <- as.numeric(x); y
[1] 10.45455
# ricordo che vengono visulizzate solo alcune cifre di quelle memorizzate:
y*1e10
[1] 104545454545
# Ecco come si potrebbero analizzare i dati in LA-4-§2 elaborati con un
# foglio di calcolo (array è una tabella; dim(3,1) specifica che
# è a 3 righe ed 1 colonna; data una tabella T l'elemento nella
# riga M e nella colonna N è indicato T[M,N])
iscr_97 <- array(c(181,146,245),dim=c(3,1))
iscr_98 <- array(c(154,126,140),dim=c(3,1))
ripe_97 <- array(c(81,38,48),dim=c(3,1))
ripe_98 <- array(c(36,35,37),dim=c(3,1))
totale <- array(c(iscr_97,rip_97,iscr_98,rip_98),dim=c(3,4)); totale
[,1] [,2] [,3] [,4]
[1,] 181 81 154 36
[2,] 146 38 126 35
[3,] 245 48 140 37
abband_1_2 <- totale[1,1]-totale[1,4]-totale[2,3]+totale[2,4]; abband_1_2
[1] 54
abband_2_3 <- totale[2,1]-totale[2,4]-totale[3,3]+totale[3,4]; abband_2_3
[1] 8
# Se voglio somme di righe o colonne posso precedere così:
sum(ripe_97)
[1] 167
sum(totale[1,])
[1] 452
sum(totale[,2])
[1] 167
# Calcoli con intervalli di indeterminazione (vedi IN-2-§4)
# moltiplicazione
xA <- 3.8; xB <- 3.9; yA <- 6.4; yB <- 6.5
p <- c(xA*yA, xA*yB, xB*yA, xB*yB); min(p); max(p)
[1] 24.32
[1] 25.35
# divisione
xA <- 1000; xB <- 1000; yA <- 920; yB <- 940
d <- c(xA/yA, xA/yB, xB/yA, xB/yB); min(d); max(d)
[1] 1.063830
[1] 1.086957
# calcolo dell'ipotenusa di un triangolo rettangolo (vedi PS-1-q.17)
c1A <- 53; c1B <- 54; c2A <- 30; c2B <- 31
ip1 <- sqrt(c1A^2+c2A^2); ip2 <- sqrt(c1B^2+c2B^2); ip1; ip2
[1] 60.90156
[1] 62.26556
# significato degli operatori logici (vedi PS-1-e4)
x<- 2; y<- 2; z<- 2; w<- 2; P<- x==y; Q<- z==w; c(!P, P&Q, P|Q)
[1] FALSE TRUE TRUE
x<- 2; y<- 2; z<- 2; w<- 1; P<- x==y; Q<- z==w; c(!P, P&Q, P|Q)
[1] FALSE FALSE TRUE
#
# >> FIGURE VARIE <<
# Alcune figure considerate in LA-4-§5
# Ecco come tracciare cerchi e quadrati vari usando il
# comando symbols
circ <- function(x,y,r,colore) symbols(x,y,circles=r,add=TRUE,fg=colore,inches = FALSE)
squa <- function(x,y,r,colore) symbols(x,y,squares=r,add=TRUE,fg=colore,inches = FALSE)
view <- c(-3,3, -3,3); orig <- c(0,0)
tx <- c(-3,-2,-1,0,1,2,3); ty <- tx
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="", asp=1)
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
circ(0,0,1,"blue"); circ(0,0,1.5,"red"); circ(-1,1,1.5,"magenta")
squa(0,0,1*2,"blue"); squa(0,0,1.5*2,"red"); squa(-1,1,1.5*2,"magenta")
#
# >> CALCOLI VARI <<
# Struttura di un termine (vedi LA-3-§2)
G <- function(x) (2^3*-x)/(9-1)+x*5; G(3); G(-5)
[1] 12
[1] -20
x <- 3; v0<-2^3; v1<- -x; v2<-v0*v1; v3<-9-1; v4<-v2/v3; v5<-x*5; v6<-v4+v5; v6
[1] 12
x <- -5; v0<-2^3; v1<- -x; v2<-v0*v1; v3<-9-1; v4<-v2/v3; v5<-x*5; v6<-v4+v5; v6
[1] -20
# Divisori e scomposizione in numeri primi (vedi LA-3-§4); floor(x) dà
# il massimo intero minore o uguale ad x; in una condizione, come quella
# che segue un if, occorre usare == invece di =.
n <- 1580; for(k in 1:n) if (n/k==floor(n/k)) print(k)
[1] 1
[1] 2
[1] 4
[1] 5
[1] 10
[1] 20
[1] 79
[1] 158
[1] 316
[1] 395
[1] 790
[1] 1580
# Occorre usare { e }quando si vuole delimitare l'esecuzione ad alcuni
# comandi; nell'esempio che segue servono per far eseguire print(k)
# e n<-n/k solo se la condizione è verificata
# while significa "fintanto che"
n<-1580; k<-2; while (k<=n) if (n/k==floor(n/k)) {print(k); n<-n/k} else k<-k+1
[1] 2
[1] 2
[1] 5
[1] 79
# La distanza dall'origine (vedi LA-3-e8) è definibile:
D <- function(x,y) sqrt(x^2+y^2)
D(1,2)
[1] 2.236068
# viene visualizzata un'approssimazione del risultato; il programma
# ne elabora in realtà una più precisa (2.236067977...), come si
# vede trasformando l'uscita in forma intera:
for (i in 8:12) print(D(1,2)*10^i)
[1] 223606798
[1] 2236067977
[1] 22360679775
[1] 223606797750
[1] 2.236068e+12
# In R le assegnazioni (vedi AL-§2) sono fatte usando <- o ->;
# occorre sempre esplicitare il simbolo di moltiplicazione, usando *
2·2
Error: unexpected input in "2·"
> 2*2
[1] 4
# Come già visto, chiamando la libreria MASS è possibile approssimare
# i numeri decimali mediante frazioni
library(MASS)
fractions(0.140140140140140140140); fractions(10.45454545454545454545)
[1] 140/999
[1] 115/11
# per passare da una forma frazionaria generata da fractions ad
# un numero usuale si può usare as.numeric:
x <- fractions(10.45454545454545454545); x
[1] 115/11
y <- as.numeric(x); y
[1] 10.45455
# ricordo che vengono visulizzate solo alcune cifre di quelle memorizzate:
y*1e10
[1] 104545454545
# Ecco come si potrebbero analizzare i dati in LA-4-§2 elaborati con un
# foglio di calcolo (array è una tabella; dim(3,1) specifica che
# è a 3 righe ed 1 colonna; data una tabella T l'elemento nella
# riga M e nella colonna N è indicato T[M,N])
iscr_97 <- array(c(181,146,245),dim=c(3,1))
iscr_98 <- array(c(154,126,140),dim=c(3,1))
ripe_97 <- array(c(81,38,48),dim=c(3,1))
ripe_98 <- array(c(36,35,37),dim=c(3,1))
totale <- array(c(iscr_97,rip_97,iscr_98,rip_98),dim=c(3,4)); totale
[,1] [,2] [,3] [,4]
[1,] 181 81 154 36
[2,] 146 38 126 35
[3,] 245 48 140 37
abband_1_2 <- totale[1,1]-totale[1,4]-totale[2,3]+totale[2,4]; abband_1_2
[1] 54
abband_2_3 <- totale[2,1]-totale[2,4]-totale[3,3]+totale[3,4]; abband_2_3
[1] 8
# Se voglio somme di righe o colonne posso precedere così:
sum(ripe_97)
[1] 167
sum(totale[1,])
[1] 452
sum(totale[,2])
[1] 167
# Calcoli con intervalli di indeterminazione (vedi IN-2-§4)
# moltiplicazione
xA <- 3.8; xB <- 3.9; yA <- 6.4; yB <- 6.5
p <- c(xA*yA, xA*yB, xB*yA, xB*yB); min(p); max(p)
[1] 24.32
[1] 25.35
# divisione
xA <- 1000; xB <- 1000; yA <- 920; yB <- 940
d <- c(xA/yA, xA/yB, xB/yA, xB/yB); min(d); max(d)
[1] 1.063830
[1] 1.086957
# calcolo dell'ipotenusa di un triangolo rettangolo (vedi PS-1-q.17)
c1A <- 53; c1B <- 54; c2A <- 30; c2B <- 31
ip1 <- sqrt(c1A^2+c2A^2); ip2 <- sqrt(c1B^2+c2B^2); ip1; ip2
[1] 60.90156
[1] 62.26556
# significato degli operatori logici (vedi PS-1-e4)
x<- 2; y<- 2; z<- 2; w<- 2; P<- x==y; Q<- z==w; c(!P, P&Q, P|Q)
[1] FALSE TRUE TRUE
x<- 2; y<- 2; z<- 2; w<- 1; P<- x==y; Q<- z==w; c(!P, P&Q, P|Q)
[1] FALSE FALSE TRUE
#
# >> FIGURE VARIE <<
# Alcune figure considerate in LA-4-§5
# Ecco come tracciare cerchi e quadrati vari usando il
# comando symbols
circ <- function(x,y,r,colore) symbols(x,y,circles=r,add=TRUE,fg=colore,inches = FALSE)
squa <- function(x,y,r,colore) symbols(x,y,squares=r,add=TRUE,fg=colore,inches = FALSE)
view <- c(-3,3, -3,3); orig <- c(0,0)
tx <- c(-3,-2,-1,0,1,2,3); ty <- tx
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="", asp=1)
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
circ(0,0,1,"blue"); circ(0,0,1.5,"red"); circ(-1,1,1.5,"magenta")
squa(0,0,1*2,"blue"); squa(0,0,1.5*2,"red"); squa(-1,1,1.5*2,"magenta")
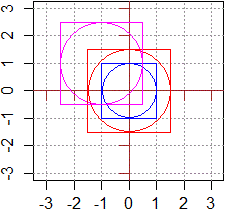 # Ecco come calcolare le coordinate del cerchio di centro (0,0)
# e raggio 1 (vedrai più avanti il significato di cos e sin)
xc <- function(angolo) cos(angolo*pi/180)
yc <- function(angolo) sin(angolo*pi/180)
c(xc(30),yc(30)); c(xc(45),yc(45)); c(xc(0),yc(0)); c(xc(180),yc(180))
[1] 0.8660254 0.5000000
[1] 0.7071068 0.7071068
[1] 1 0
[1] -1.000000e+00 1.224606e-16 (è circa -1 0)
# ed ecco come tracciare tale cerchio in modo alternativo al precedente
view <- c(-3,3, -3,3); orig <- c(0,0)
tx <- c(-3,-2,-1,0,1,2,3); ty <- tx
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="", asp=1)
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
ang <- seq(0, 360, 360/1000)
lines(xc(ang), yc(ang))
# alcuni punti su di esso
points(xc(30),yc(30)); points(xc(-45),yc(-45)); points(xc(240),yc(240))
# alcune trasformazioni di esso
lines(xc(ang)+2,yc(ang)-1)
lines(xc(ang)/2+2,yc(ang)*2-1,col="red")
# ed un esagono regolare
ang1 <- seq(0, 360, 360/6); lines(xc(ang1)*2,yc(ang1)*2,col="blue")
# Ecco come calcolare le coordinate del cerchio di centro (0,0)
# e raggio 1 (vedrai più avanti il significato di cos e sin)
xc <- function(angolo) cos(angolo*pi/180)
yc <- function(angolo) sin(angolo*pi/180)
c(xc(30),yc(30)); c(xc(45),yc(45)); c(xc(0),yc(0)); c(xc(180),yc(180))
[1] 0.8660254 0.5000000
[1] 0.7071068 0.7071068
[1] 1 0
[1] -1.000000e+00 1.224606e-16 (è circa -1 0)
# ed ecco come tracciare tale cerchio in modo alternativo al precedente
view <- c(-3,3, -3,3); orig <- c(0,0)
tx <- c(-3,-2,-1,0,1,2,3); ty <- tx
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="", asp=1)
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
ang <- seq(0, 360, 360/1000)
lines(xc(ang), yc(ang))
# alcuni punti su di esso
points(xc(30),yc(30)); points(xc(-45),yc(-45)); points(xc(240),yc(240))
# alcune trasformazioni di esso
lines(xc(ang)+2,yc(ang)-1)
lines(xc(ang)/2+2,yc(ang)*2-1,col="red")
# ed un esagono regolare
ang1 <- seq(0, 360, 360/6); lines(xc(ang1)*2,yc(ang1)*2,col="blue")
 # La croce visualizzata è stata ottenuta (temporameante) cliccando
# sul punto indicato dopo aver scritto locator(1); in uscita si hanno
# le coordinate approssimative del punto cliccato.
locator(1)
$x
[1] -0.5211999
$y
[1] 0.5136463
# Volendo posso memorizzare le cordinate del punto cliccato aggiungendo
# $x e $y, come si vede qui sotto:
loc <- locator(1); xp <- loc$x; yp <- loc$y; xp; yp
[1] -0.5211999
[1] 0.5136463
# locator può essere usato anche per trovare graficamente soluzioni
# di equazioni; ecco ad es. come risolvere questo esercizio:
ty <- c(0,1,2,3,4,5,6,7,8,9,10)*10
tx <- c(0,1,2,3,4,5,6,7,8,9,10,11,12,13)
tx <- tx*10; k <- 100/124.2
f <- function(x) x*k
view <- c(0,130,0,100); orig <- c(0,0)
# i soliti comandi:
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
# il grafico di f e l'evidenziazione del punto (124.2, 100)
curve(f, add=TRUE, col="blue", from=0)
lines(c(124.2,124.2,0),c(0,100,100))
locator(1)
$x
[1] 34.41743
$y
[1] 27.96164
# La croce visualizzata è stata ottenuta (temporameante) cliccando
# sul punto indicato dopo aver scritto locator(1); in uscita si hanno
# le coordinate approssimative del punto cliccato.
locator(1)
$x
[1] -0.5211999
$y
[1] 0.5136463
# Volendo posso memorizzare le cordinate del punto cliccato aggiungendo
# $x e $y, come si vede qui sotto:
loc <- locator(1); xp <- loc$x; yp <- loc$y; xp; yp
[1] -0.5211999
[1] 0.5136463
# locator può essere usato anche per trovare graficamente soluzioni
# di equazioni; ecco ad es. come risolvere questo esercizio:
ty <- c(0,1,2,3,4,5,6,7,8,9,10)*10
tx <- c(0,1,2,3,4,5,6,7,8,9,10,11,12,13)
tx <- tx*10; k <- 100/124.2
f <- function(x) x*k
view <- c(0,130,0,100); orig <- c(0,0)
# i soliti comandi:
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
# il grafico di f e l'evidenziazione del punto (124.2, 100)
curve(f, add=TRUE, col="blue", from=0)
lines(c(124.2,124.2,0),c(0,100,100))
locator(1)
$x
[1] 34.41743
$y
[1] 27.96164
 # la figura a destra corrisponde al seguente "zoom" (con tacche
# più fitte):
view <- c(160,170,35,45)
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
curve(f, add=TRUE, col="blue", from=0)
ttx <- 160+tx/10
abline(v=ttx,col="orange",lty=3)
tty <- 35+ty/10
abline(h=tty,col="orange",lty=3)
locator(1)
$x
[1] 34.1082
$y
[1] 27.49286
# Concludo che al 2.75% corrispondono 3.4 miliardi. Ovviamente in questo
# caso è facile procedere numericamente: da x*k = 2.75 ho x = ...
2.75/k
[1] 3.4155
#
# Come ottenere altri poligoni regolari (vedi LA-4-e12) con comandi analoghi
# (xc e yc per avere le coordinate di un punto P del cerchio dato l'angolo
# in gradi di OP)
view <- c(-1,1, -1,1); orig <- c(0,0)
tx <- c(-1,-0.5,0,0.5,1); ty <- tx
abline(h = ty,col="grey"); abline(v = tx, col="grey")
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="", asp=1)
abline(h = ty,col="grey"); abline(v = tx, col="grey")
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
xc <- function(angolo) cos(angolo*pi/180); yc <- function(angolo) sin(angolo*pi/180)
ang <- function(n) seq(0, 360, 360/n)
for (n in 2:8) lines(xc(ang(n)),yc(ang(n)),col=n)
# la figura a destra corrisponde al seguente "zoom" (con tacche
# più fitte):
view <- c(160,170,35,45)
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
curve(f, add=TRUE, col="blue", from=0)
ttx <- 160+tx/10
abline(v=ttx,col="orange",lty=3)
tty <- 35+ty/10
abline(h=tty,col="orange",lty=3)
locator(1)
$x
[1] 34.1082
$y
[1] 27.49286
# Concludo che al 2.75% corrispondono 3.4 miliardi. Ovviamente in questo
# caso è facile procedere numericamente: da x*k = 2.75 ho x = ...
2.75/k
[1] 3.4155
#
# Come ottenere altri poligoni regolari (vedi LA-4-e12) con comandi analoghi
# (xc e yc per avere le coordinate di un punto P del cerchio dato l'angolo
# in gradi di OP)
view <- c(-1,1, -1,1); orig <- c(0,0)
tx <- c(-1,-0.5,0,0.5,1); ty <- tx
abline(h = ty,col="grey"); abline(v = tx, col="grey")
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="", asp=1)
abline(h = ty,col="grey"); abline(v = tx, col="grey")
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
xc <- function(angolo) cos(angolo*pi/180); yc <- function(angolo) sin(angolo*pi/180)
ang <- function(n) seq(0, 360, 360/n)
for (n in 2:8) lines(xc(ang(n)),yc(ang(n)),col=n)
 # un poligono qualunque si può tracciare col comando polygon (si
# mettono le x e le y in due collezioni di numeri:
view <- c(-3,3, -3,3); orig <- c(0,0)
tx <- c(-3,-2,-1,0,1,2,3); ty <- tx
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="", asp=1)
abline(h = ty,col="grey"); abline(v = tx, col="grey")
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
x <- c(0,1,2,2.5,-2); y <- c(1,3,-1.6,-2,-1)
polygon(x, y, col="yellow", border="blue")
# un poligono qualunque si può tracciare col comando polygon (si
# mettono le x e le y in due collezioni di numeri:
view <- c(-3,3, -3,3); orig <- c(0,0)
tx <- c(-3,-2,-1,0,1,2,3); ty <- tx
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="", asp=1)
abline(h = ty,col="grey"); abline(v = tx, col="grey")
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
x <- c(0,1,2,2.5,-2); y <- c(1,3,-1.6,-2,-1)
polygon(x, y, col="yellow", border="blue")
 #
# figure mediante caratteri (vedi PS-1-e8); text(x,y,"scritta") scrive
# la "scritta" centrata nel punto (x,y) della finestra grafica
view <- c(0,11, 0,11)
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="", asp=1)
for(i in 1:10) text(i,9,"O")
for(i in 9:5) text(1,i,"O")
for(i in 9:5) text(10,i,"O")
for(i in 1:10) text(i,4,"O")
#
# figure mediante caratteri (vedi PS-1-e8); text(x,y,"scritta") scrive
# la "scritta" centrata nel punto (x,y) della finestra grafica
view <- c(0,11, 0,11)
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="", asp=1)
for(i in 1:10) text(i,9,"O")
for(i in 9:5) text(1,i,"O")
for(i in 9:5) text(10,i,"O")
for(i in 1:10) text(i,4,"O")
 for(i in 10:5) text(1,i,"O")
for(i in 1:6) text(i,5,"O")
for(i in 1:6) text(i,11-i,"O")
#
# corona circolare (vedi MS-1-e7)
view <- c(-6,6, -6,6); orig <- c(0,0)
tx <- c(0,2,4,6,-2,-4,-6); ty <- tx
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="", asp=1)
abline(h = ty,col="grey"); abline(v = tx, col="grey")
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
fxy <- function(x,y) sqrt(x*x+y*y)
x1 <- -5; x2 <- 5; y1 <- -5; y2 <- 5
dx <- (x2-x1)/100; dy <- (y2-y1)/100; I <- 0:100; J <- 0:100
for(i in I) for(j in J) {x<-x1+dx*i;y<-y1+dy*j;if(fxy(x,y)<=5 & fxy(x,y)>=3) points(x,y,pch=".",col="blue")}
for(i in 10:5) text(1,i,"O")
for(i in 1:6) text(i,5,"O")
for(i in 1:6) text(i,11-i,"O")
#
# corona circolare (vedi MS-1-e7)
view <- c(-6,6, -6,6); orig <- c(0,0)
tx <- c(0,2,4,6,-2,-4,-6); ty <- tx
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="", asp=1)
abline(h = ty,col="grey"); abline(v = tx, col="grey")
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
fxy <- function(x,y) sqrt(x*x+y*y)
x1 <- -5; x2 <- 5; y1 <- -5; y2 <- 5
dx <- (x2-x1)/100; dy <- (y2-y1)/100; I <- 0:100; J <- 0:100
for(i in I) for(j in J) {x<-x1+dx*i;y<-y1+dy*j;if(fxy(x,y)<=5 & fxy(x,y)>=3) points(x,y,pch=".",col="blue")}
 #
# figure tridimensionali (vedi MS-1-e10)
# Ecco, a scatola nera, come realizzare il parallelepipedo dell'es.:
# in x ed y metto le coordinate di un rettangolo della faccia e in
# z metto una tabella 2*2 con le corrispondenti quote;
# nel comando persp indico gli intervalli per le x, le y e le z
# da rappresentare; ottengo la figura sotto a sinistra.
u<- c(3,3,3,3); z<- array(u,dim=c(2,2)); x<- c(6,8); y<- c(0,3)
PR <- persp(x,y,z,zlim=c(0,10),xlim=c(0,8),ylim=c(0,6),border="blue")
#
# figure tridimensionali (vedi MS-1-e10)
# Ecco, a scatola nera, come realizzare il parallelepipedo dell'es.:
# in x ed y metto le coordinate di un rettangolo della faccia e in
# z metto una tabella 2*2 con le corrispondenti quote;
# nel comando persp indico gli intervalli per le x, le y e le z
# da rappresentare; ottengo la figura sotto a sinistra.
u<- c(3,3,3,3); z<- array(u,dim=c(2,2)); x<- c(6,8); y<- c(0,3)
PR <- persp(x,y,z,zlim=c(0,10),xlim=c(0,8),ylim=c(0,6),border="blue")
 # poi aggiungo (nella stessa visione prospettica, che ho chiamato PR)
# col comando trans3d le coordinate delle altre facce
lines(trans3d(c(8,8,6,6,8),c(0,3,3,0,0),c(7,7,7,7,7),pmat=PR),col="red")
lines(trans3d(c(6,6),c(0,0),c(3,7),pmat=PR),col="green4")
lines(trans3d(c(6,6),c(3,3),c(3,7),pmat=PR),col="orange")
lines(trans3d(c(8,8),c(3,3),c(3,7),pmat=PR),col="brown")
#
# >> APPROFONDIMENTI <<
# Volendo, fra il "plot" e l'"abline" dei comandi in viola posso
# mettere comandi che mi infittiscono la griglia, ad es.
for (oriz in -3:25) abline(h=oriz,col="yellow")
for (vert in (-5:30)*1/5) abline(v=vert,col="yellow")
# poi aggiungo (nella stessa visione prospettica, che ho chiamato PR)
# col comando trans3d le coordinate delle altre facce
lines(trans3d(c(8,8,6,6,8),c(0,3,3,0,0),c(7,7,7,7,7),pmat=PR),col="red")
lines(trans3d(c(6,6),c(0,0),c(3,7),pmat=PR),col="green4")
lines(trans3d(c(6,6),c(3,3),c(3,7),pmat=PR),col="orange")
lines(trans3d(c(8,8),c(3,3),c(3,7),pmat=PR),col="brown")
#
# >> APPROFONDIMENTI <<
# Volendo, fra il "plot" e l'"abline" dei comandi in viola posso
# mettere comandi che mi infittiscono la griglia, ad es.
for (oriz in -3:25) abline(h=oriz,col="yellow")
for (vert in (-5:30)*1/5) abline(v=vert,col="yellow")
 # (-5:30)*1/5 sta per da -1 a 6 con passo 1/5; altro es.: per
# da 200 a 500 passo 50: 200/50=4, 500/50=10 quindi (4:10)*50
#
# Oppure mettere nel modo seguente gli estremi in cui tracciare
# le tacche e l'ampiezza di esse, e il colore:
ttx <- c(-3,25,1); tty <- c(-1,6,0.2); color="yellow"
# e quindi:
ttxx <- ttx/ttx[3]; ttyy <- tty/tty[3]
for (oriz in (ttxx[1]:ttxx[2])*ttx[3]) abline(h=oriz,col=color)
for (vert in (ttyy[1]:ttyy[2])*tty[3]) abline(v=vert,col=color)
#
# In definita posso mettere:
# descrizione delle funzioni
f <- function(x) 1.45*x; g <- function(x) 4.5*x
# rettangolo cartesiano e origine degli assi
view <- c(-1,6,-3,25); orig <- c(0,0)
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
# inizio, fine, ampiezza delle tacche
ttx <- c(-3,25, 1); tty <- c(-1,6, 0.2); color="yellow"
ttxx <- ttx/ttx[3]; ttyy <- tty/tty[3]
for (oriz in (ttxx[1]:ttxx[2])*ttx[3]) abline(h=oriz,col=color)
for (vert in (ttyy[1]:ttyy[2])*tty[3]) abline(v=vert,col=color)
ttx <- c(0,25, 5); tty <- c(-1,6, 1); color="grey"
ttxx <- ttx/ttx[3]; ttyy <- tty/tty[3]
for (oriz in (ttxx[1]:ttxx[2])*ttx[3]) abline(h=oriz,col=color)
for (vert in (ttyy[1]:ttyy[2])*tty[3]) abline(v=vert,col=color)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
# rappresentazione delle funzioni
curve(g, add=TRUE,col="green4", from=0, to=5)
curve(f, add=TRUE,col="blue", from=0, to=5)
# (-5:30)*1/5 sta per da -1 a 6 con passo 1/5; altro es.: per
# da 200 a 500 passo 50: 200/50=4, 500/50=10 quindi (4:10)*50
#
# Oppure mettere nel modo seguente gli estremi in cui tracciare
# le tacche e l'ampiezza di esse, e il colore:
ttx <- c(-3,25,1); tty <- c(-1,6,0.2); color="yellow"
# e quindi:
ttxx <- ttx/ttx[3]; ttyy <- tty/tty[3]
for (oriz in (ttxx[1]:ttxx[2])*ttx[3]) abline(h=oriz,col=color)
for (vert in (ttyy[1]:ttyy[2])*tty[3]) abline(v=vert,col=color)
#
# In definita posso mettere:
# descrizione delle funzioni
f <- function(x) 1.45*x; g <- function(x) 4.5*x
# rettangolo cartesiano e origine degli assi
view <- c(-1,6,-3,25); orig <- c(0,0)
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
# inizio, fine, ampiezza delle tacche
ttx <- c(-3,25, 1); tty <- c(-1,6, 0.2); color="yellow"
ttxx <- ttx/ttx[3]; ttyy <- tty/tty[3]
for (oriz in (ttxx[1]:ttxx[2])*ttx[3]) abline(h=oriz,col=color)
for (vert in (ttyy[1]:ttyy[2])*tty[3]) abline(v=vert,col=color)
ttx <- c(0,25, 5); tty <- c(-1,6, 1); color="grey"
ttxx <- ttx/ttx[3]; ttyy <- tty/tty[3]
for (oriz in (ttxx[1]:ttxx[2])*ttx[3]) abline(h=oriz,col=color)
for (vert in (ttyy[1]:ttyy[2])*tty[3]) abline(v=vert,col=color)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
# rappresentazione delle funzioni
curve(g, add=TRUE,col="green4", from=0, to=5)
curve(f, add=TRUE,col="blue", from=0, to=5)


 # La distribuzione percentuale degli stessi dati
sum(dati); datiperc <- dati/sum(dati)*100; datiperc
etichette <- c("al","tb","v","ab","tr","a")
barplot(datiperc, names.arg=etichette, ylim=c(0,40))
abline(h=c(0,10,20,30,40),col="red",lty=3)
[1] 434100
[1] 26.756047 2.143746 8.236812 10.421101 13.572679 38.869615
# La distribuzione percentuale degli stessi dati
sum(dati); datiperc <- dati/sum(dati)*100; datiperc
etichette <- c("al","tb","v","ab","tr","a")
barplot(datiperc, names.arg=etichette, ylim=c(0,40))
abline(h=c(0,10,20,30,40),col="red",lty=3)
[1] 434100
[1] 26.756047 2.143746 8.236812 10.421101 13.572679 38.869615
 # Ecco una rappresentazione con (vedi LS-1-§4) un diagramma a striscia
barplot(height=cbind(dati/sum(dati)*100),horiz=TRUE)
# Ecco una rappresentazione con (vedi LS-1-§4) un diagramma a striscia
barplot(height=cbind(dati/sum(dati)*100),horiz=TRUE)
 # e con un diagrammi a settori circolari (o a torta - "pie" in inglese)
pie(dati, labels=etichette)
# e con un diagrammi a settori circolari (o a torta - "pie" in inglese)
pie(dati, labels=etichette)
 dati/sum(dati)*360 # ampiezza in gradi dei settori
[1] 96.321769 7.717484 29.652522 37.515964 48.861645 139.930615
# L'areogramma soprastante a destra è stato ottenuto (usando paste,
# ossia "incolla") con:
percentuali <- round(dati/sum(dati)*1000)/10
pie(dati, labels=paste(etichette,":",percentuali,"%"))
# round arrotonda agli interi; quindi round(x*1000)/1000 arrotonda
# ai millesimi: 0.268, 0.021,...,0.389 e round(x*1000)/10 moltiplica
# tali valori per 100: 2.68, 0.21,...,3.89.
#
# >> FINESTRE e IMMAGINI <<
# Se vogliamo dimensionare in modi particolari i grafici, in modo da
# confrontarli, possiamo dimensionare le finestre grafiche col comando
# windows; ad es. windows(3,3) apre una nuova finestra di 3*3 pollici
# in cui viene tracciato il successivo grafico. Questo può essere comodo
# anche per unire e sovrapporre grafici fatti in tempi diversi.
# Ecco come conviene salvare un'immagine. Si clicca su di essa e, dal menu
# che si apre cliccando, la si salva come BitMap e la si incolla su Paint
# (o un altro programma di grafica), si aggiunge quello che si vuole e la si
# ridimensiona. A questo punto o la si copia e salva nel documento in cui si
# sta operando o, da Paint, la si salva in formato GIF, che occupa poco spazio
# e consente di operare successive modifiche con Paint.
#
# >> FUNZIONI <<
# Esempi di funzioni ad 1, 2 e 3 input (vedi LS-2-§3)
perc1 <- function(dato) dato/12450*100
perc2 <- function(dato,totale) dato/totale*100
ripart <- function(dato,totale,base) dato/totale*base
perc1(5400); perc2(5400,12450); ripart(5400,12450,100); ripart(5400,12450,360)
[1] 43.37349
[1] 43.37349
[1] 43.37349
[1] 156.1446
# un altro esempio
k <- 4; f <- function(x) k*x^2; f(3)
[1] 36
# Possiamo comporre più funzioni (sqrt indica la radice quadrata):
F1 <- function(x) x^2; F2 <- function(x) sqrt(x)
F1(F2(5)); F2(F1(5)); F1(F2(-5)); F2(F1(-5))
[1] 5
[1] 5
[1] NaN
Warning message: In sqrt(x) : NaNs produced
[1] 5
#
# >> GRAFICI DI FUNZIONI di 1 variabile <<
# Un modo semplice per tracciare il grafico di una funzione (vedi LS-2-§3),
# usando plot; volendo posso aggiungere abline che (come già visto)
# traccia rette x = h e y = v
F <- function(x) x*360/434100
plot(F, 0,400E3, col="blue")
abline(h=0, col="brown"); abline(v=0, col="brown")
dati/sum(dati)*360 # ampiezza in gradi dei settori
[1] 96.321769 7.717484 29.652522 37.515964 48.861645 139.930615
# L'areogramma soprastante a destra è stato ottenuto (usando paste,
# ossia "incolla") con:
percentuali <- round(dati/sum(dati)*1000)/10
pie(dati, labels=paste(etichette,":",percentuali,"%"))
# round arrotonda agli interi; quindi round(x*1000)/1000 arrotonda
# ai millesimi: 0.268, 0.021,...,0.389 e round(x*1000)/10 moltiplica
# tali valori per 100: 2.68, 0.21,...,3.89.
#
# >> FINESTRE e IMMAGINI <<
# Se vogliamo dimensionare in modi particolari i grafici, in modo da
# confrontarli, possiamo dimensionare le finestre grafiche col comando
# windows; ad es. windows(3,3) apre una nuova finestra di 3*3 pollici
# in cui viene tracciato il successivo grafico. Questo può essere comodo
# anche per unire e sovrapporre grafici fatti in tempi diversi.
# Ecco come conviene salvare un'immagine. Si clicca su di essa e, dal menu
# che si apre cliccando, la si salva come BitMap e la si incolla su Paint
# (o un altro programma di grafica), si aggiunge quello che si vuole e la si
# ridimensiona. A questo punto o la si copia e salva nel documento in cui si
# sta operando o, da Paint, la si salva in formato GIF, che occupa poco spazio
# e consente di operare successive modifiche con Paint.
#
# >> FUNZIONI <<
# Esempi di funzioni ad 1, 2 e 3 input (vedi LS-2-§3)
perc1 <- function(dato) dato/12450*100
perc2 <- function(dato,totale) dato/totale*100
ripart <- function(dato,totale,base) dato/totale*base
perc1(5400); perc2(5400,12450); ripart(5400,12450,100); ripart(5400,12450,360)
[1] 43.37349
[1] 43.37349
[1] 43.37349
[1] 156.1446
# un altro esempio
k <- 4; f <- function(x) k*x^2; f(3)
[1] 36
# Possiamo comporre più funzioni (sqrt indica la radice quadrata):
F1 <- function(x) x^2; F2 <- function(x) sqrt(x)
F1(F2(5)); F2(F1(5)); F1(F2(-5)); F2(F1(-5))
[1] 5
[1] 5
[1] NaN
Warning message: In sqrt(x) : NaNs produced
[1] 5
#
# >> GRAFICI DI FUNZIONI di 1 variabile <<
# Un modo semplice per tracciare il grafico di una funzione (vedi LS-2-§3),
# usando plot; volendo posso aggiungere abline che (come già visto)
# traccia rette x = h e y = v
F <- function(x) x*360/434100
plot(F, 0,400E3, col="blue")
abline(h=0, col="brown"); abline(v=0, col="brown")
 # Un modo per ottenere un grafico migliore (e aggiungerne altri
# sulla stessa scala) è il seguente; si scelgono la parte di piano
# cartesiano [x1,x2]*[y1,y2] assegnandone i valori ad una variabile
# (ad es. di nome view), l'origine (assegnandone le coordinate ad una
# variabile (ad es. orig), e le posizioni delle tacche sugli assi
# attraverso cui far passare la griglia ad altre due (ad es. tx e ty)
view <- c(-10e3,400e3,-10,300); orig <- c(0,0)
tx <- c(0,50,100,150,200,250,300,350,400)*1000
ty <- c(0,50,100,150,200,250,300)
# poi si copiano e incollano i seguenti comandi
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
curve(F, add=TRUE, col="blue", from=0, to=400e3)
# se nell'ultimo comando, "curve", non metto "from" e "to" il grafico
# viene disegnato su tutto l'intervallo scelto per le ascisse
# Un modo per ottenere un grafico migliore (e aggiungerne altri
# sulla stessa scala) è il seguente; si scelgono la parte di piano
# cartesiano [x1,x2]*[y1,y2] assegnandone i valori ad una variabile
# (ad es. di nome view), l'origine (assegnandone le coordinate ad una
# variabile (ad es. orig), e le posizioni delle tacche sugli assi
# attraverso cui far passare la griglia ad altre due (ad es. tx e ty)
view <- c(-10e3,400e3,-10,300); orig <- c(0,0)
tx <- c(0,50,100,150,200,250,300,350,400)*1000
ty <- c(0,50,100,150,200,250,300)
# poi si copiano e incollano i seguenti comandi
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
curve(F, add=TRUE, col="blue", from=0, to=400e3)
# se nell'ultimo comando, "curve", non metto "from" e "to" il grafico
# viene disegnato su tutto l'intervallo scelto per le ascisse
 # Con alcune opzioni è possibile decidere (con "n") il numero di
# punti, decidere se isolarli (con "type"), darne forma ("pch")
# e dimensioni ("cex"). Due esempi:
curve(F,add=TRUE,col="blue",from=0,to=400e3, type="p", n=50)
curve(F,add=TRUE,col="blue",from=0,to=400e3, type="p", n=50, pch=".", cex=3)
# Con alcune opzioni è possibile decidere (con "n") il numero di
# punti, decidere se isolarli (con "type"), darne forma ("pch")
# e dimensioni ("cex"). Due esempi:
curve(F,add=TRUE,col="blue",from=0,to=400e3, type="p", n=50)
curve(F,add=TRUE,col="blue",from=0,to=400e3, type="p", n=50, pch=".", cex=3)
 # Cambiando le scale posso rappresentare altre funzioni:
f <- function(x) 1.45*x; g <- function(x) 4.5*x
view <- c(-1,6,-3,25); orig <- c(0,0)
ty <- c(0,5,10,15,20,25)
tx <- c(-1,0,1,2,3,4,5,6)
# faccio seguire i comandi nelle righe sopra colorate in viola
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
# e poi (add=TRUE serve per poter aggiungere grafici):
curve(g, add=TRUE,col="green4", from=0, to=5)
curve(f, add=TRUE,col="blue", from=0, to=5)
# Cambiando le scale posso rappresentare altre funzioni:
f <- function(x) 1.45*x; g <- function(x) 4.5*x
view <- c(-1,6,-3,25); orig <- c(0,0)
ty <- c(0,5,10,15,20,25)
tx <- c(-1,0,1,2,3,4,5,6)
# faccio seguire i comandi nelle righe sopra colorate in viola
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
# e poi (add=TRUE serve per poter aggiungere grafici):
curve(g, add=TRUE,col="green4", from=0, to=5)
curve(f, add=TRUE,col="blue", from=0, to=5)
 #
# >> GRAFICI per PUNTI <<
# Se voglio rappresentare le seguenti coppie di punti (vedi LS-2-q.18):
# 78, 212
# 79, 226
# 80, 212
# 81, 217
# 82, 283
# metto in x i primi elementi e in y i secondi, scelgo
# la porzione di piano e le tacche:
x <- c(78,79,80,81,82); y <- c(212,226,212,217,283)
view <- c(75,85, -50,300); orig <- c(75,0)
tx <- c(75,76,77,78,79,80,81,82,83,84,85)
ty <- c(-50,0,50,100,150,200,250,300)
# quindi attacco i soliti comandi:
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
# e, infine, uso il comando lines per tracciare una poligonale
# da (78,212) a (79,226) ... a (82,283):
lines(x,y)
#
# >> GRAFICI per PUNTI <<
# Se voglio rappresentare le seguenti coppie di punti (vedi LS-2-q.18):
# 78, 212
# 79, 226
# 80, 212
# 81, 217
# 82, 283
# metto in x i primi elementi e in y i secondi, scelgo
# la porzione di piano e le tacche:
x <- c(78,79,80,81,82); y <- c(212,226,212,217,283)
view <- c(75,85, -50,300); orig <- c(75,0)
tx <- c(75,76,77,78,79,80,81,82,83,84,85)
ty <- c(-50,0,50,100,150,200,250,300)
# quindi attacco i soliti comandi:
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
# e, infine, uso il comando lines per tracciare una poligonale
# da (78,212) a (79,226) ... a (82,283):
lines(x,y)
 # cambiando le righe iniziali ho il grafico sopra a destra:
view <- c(77,83, 200,300); orig <- c(77,200)
tx <- c(77,78,79,80,81,82,83)
ty <- c(200,210,220,230,240,250,260,270,280,290,300)
#
# Un altro esempio (vedi LS-2-e3)
# Mettiamo i punti da rappresentare, assi e tacche:
x <- c(7+55/60, 9+36/60, 9+40/60, 10+41/60, 10+50/60, 12+43/60)
y <- c(0, 219, 219, 316, 316, 626)
view <- c(7,13, -20,650); orig <- c(7,0)
tx <- c(7,8,9,10,11,12,13); ty <- c(0,100,200,300,400,500,600)
# quindi attacchiamo i soliti comandi:
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
# e, infine, oltre a lines, il comando points per evidenziare i singoli
# punti (points traccia punti, senza opzioni mediante dei circoletti,
# altrimenti in svariate forme; con pch="." comandiamo di tracciarli come
# punti tipografici, con cex=4 diamo le dimensioni di essi, con col diamo
# loro il colore)
lines(x,y); points(x,y, pch=".", cex=4, col="blue")
# cambiando le righe iniziali ho il grafico sopra a destra:
view <- c(77,83, 200,300); orig <- c(77,200)
tx <- c(77,78,79,80,81,82,83)
ty <- c(200,210,220,230,240,250,260,270,280,290,300)
#
# Un altro esempio (vedi LS-2-e3)
# Mettiamo i punti da rappresentare, assi e tacche:
x <- c(7+55/60, 9+36/60, 9+40/60, 10+41/60, 10+50/60, 12+43/60)
y <- c(0, 219, 219, 316, 316, 626)
view <- c(7,13, -20,650); orig <- c(7,0)
tx <- c(7,8,9,10,11,12,13); ty <- c(0,100,200,300,400,500,600)
# quindi attacchiamo i soliti comandi:
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
# e, infine, oltre a lines, il comando points per evidenziare i singoli
# punti (points traccia punti, senza opzioni mediante dei circoletti,
# altrimenti in svariate forme; con pch="." comandiamo di tracciarli come
# punti tipografici, con cex=4 diamo le dimensioni di essi, con col diamo
# loro il colore)
lines(x,y); points(x,y, pch=".", cex=4, col="blue")
 #
# Se (vedi LS-2-e20) voglio andare a capo durante la scrittura di un
# comando basta che batta a capo: il comando viene spezzato su più
# righe e viene interpretato correttamente da R
x <- c(0.5,-0.5,-0.5, 1, 1,-1,-1, 1.5, 1.5,-1.5, # continua nella riga successiva
-1.5, 2, 2,-2,-2, 2.5, 2.5)
y <- c(0.5, 0.5,-0.5,-0.5, 1, 1,-1,-1, 1.5, 1.5, # continua
-1.5,-1.5, 2, 2,-2,-2, 2.5)
# Un'alternativa, per caricare molti dati in una variabile, e'
# caricarne una parte e poi aggiungere i rimanenti alla stessa
# variabile con un'altra assegnazione:
x <- c(0.5,-0.5,-0.5, 1, 1,-1,-1, 1.5, 1.5,-1.5)
x <- c(x ,-1.5, 2, 2,-2,-2, 2.5, 2.5)
view <- c(-4,4, -4,4); orig <- c(0,0)
tx <- c(-4,-3,-2,-1,0,1,2,3,4); ty <- tx
# Con asp possiamo dare il rapporto Δy/Δx usato nel grafico;
# con asp=1 otteniamo un sistema monometrico
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="",asp=1)
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
lines(x,y)
#
# Se (vedi LS-2-e20) voglio andare a capo durante la scrittura di un
# comando basta che batta a capo: il comando viene spezzato su più
# righe e viene interpretato correttamente da R
x <- c(0.5,-0.5,-0.5, 1, 1,-1,-1, 1.5, 1.5,-1.5, # continua nella riga successiva
-1.5, 2, 2,-2,-2, 2.5, 2.5)
y <- c(0.5, 0.5,-0.5,-0.5, 1, 1,-1,-1, 1.5, 1.5, # continua
-1.5,-1.5, 2, 2,-2,-2, 2.5)
# Un'alternativa, per caricare molti dati in una variabile, e'
# caricarne una parte e poi aggiungere i rimanenti alla stessa
# variabile con un'altra assegnazione:
x <- c(0.5,-0.5,-0.5, 1, 1,-1,-1, 1.5, 1.5,-1.5)
x <- c(x ,-1.5, 2, 2,-2,-2, 2.5, 2.5)
view <- c(-4,4, -4,4); orig <- c(0,0)
tx <- c(-4,-3,-2,-1,0,1,2,3,4); ty <- tx
# Con asp possiamo dare il rapporto Δy/Δx usato nel grafico;
# con asp=1 otteniamo un sistema monometrico
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="",asp=1)
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
lines(x,y)
 # Vediamo come caricare il file con i dati sul salto in alto (vedi LS-2-§2);
# prima ne esamino le righe iniziali, per controllare che cos'è;
# Uso il comando readLines
readLines("http://macosa.dima.unige.it/om/prg/gfu/altomas.txt", n=5)
[1] "'record alto maschi dal 32 ogni 4 anni" "1932,203"
[3] "1936,207" "1940,209"
[5] "1944,211"
# ok; è una tabella di dati; ogni riga contiene due dati separati
# da una virgola; il comando per leggere la tabella è read.table;
# salto (con skip) la prima riga di commenti e uso "," come separatore;
# vedo le prime tre righe
read.table("http://macosa.dima.unige.it/om/prg/gfu/altomas.txt",sep=",",nrows=3,skip=1)
V1 V2
1 1932 203
2 1936 207
3 1940 209
# se voglio vedere tutto faccio:
read.table("http://macosa.dima.unige.it/om/prg/gfu/altomas.txt",sep=",",skip=1)
V1 V2
1 1932 203
2 1936 207
...
19 2004 245
20 2008 245
# Come vedo, le due variabili vengono chiamate V1 e V2. Metto
# in hm e in hf la tabella dei maschi e quella delle femmine (che
# è in un altro file che potrei esaminare allo stesso modo):
hm <- read.table("http://macosa.dima.unige.it/om/prg/gfu/altomas.txt",sep=",",skip=1)
hf <- read.table("http://macosa.dima.unige.it/om/prg/gfu/altofem.txt",sep=",",skip=1)
# il grafico per punti dei primi dati lo posso ottenre così (hm$V1 contiene
# i valori di V1 della tabella hm; hm$V2 contiene ...)
plot(hm$V1,hm$V2)
# Vediamo come caricare il file con i dati sul salto in alto (vedi LS-2-§2);
# prima ne esamino le righe iniziali, per controllare che cos'è;
# Uso il comando readLines
readLines("http://macosa.dima.unige.it/om/prg/gfu/altomas.txt", n=5)
[1] "'record alto maschi dal 32 ogni 4 anni" "1932,203"
[3] "1936,207" "1940,209"
[5] "1944,211"
# ok; è una tabella di dati; ogni riga contiene due dati separati
# da una virgola; il comando per leggere la tabella è read.table;
# salto (con skip) la prima riga di commenti e uso "," come separatore;
# vedo le prime tre righe
read.table("http://macosa.dima.unige.it/om/prg/gfu/altomas.txt",sep=",",nrows=3,skip=1)
V1 V2
1 1932 203
2 1936 207
3 1940 209
# se voglio vedere tutto faccio:
read.table("http://macosa.dima.unige.it/om/prg/gfu/altomas.txt",sep=",",skip=1)
V1 V2
1 1932 203
2 1936 207
...
19 2004 245
20 2008 245
# Come vedo, le due variabili vengono chiamate V1 e V2. Metto
# in hm e in hf la tabella dei maschi e quella delle femmine (che
# è in un altro file che potrei esaminare allo stesso modo):
hm <- read.table("http://macosa.dima.unige.it/om/prg/gfu/altomas.txt",sep=",",skip=1)
hf <- read.table("http://macosa.dima.unige.it/om/prg/gfu/altofem.txt",sep=",",skip=1)
# il grafico per punti dei primi dati lo posso ottenre così (hm$V1 contiene
# i valori di V1 della tabella hm; hm$V2 contiene ...)
plot(hm$V1,hm$V2)
 # per rappresentare meglio i dati procedo nel solito modo:
tx <- 1900+c(3,4,5,6,7,8,9,10,11)*10
ty <- 160+c(0,1,2,3,4,5,6,7,8,9,10)*10
view <- c(1930,2010, 160,250); orig <- c(1930,160)
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
lines(hm$V1,hm$V2); lines(hf$V1,hf$V2)
points(hm$V1,hm$V2, pch=".", cex=4, col="blue")
points(hf$V1,hf$V2, pch=".", cex=4, col="red")
# per rappresentare meglio i dati procedo nel solito modo:
tx <- 1900+c(3,4,5,6,7,8,9,10,11)*10
ty <- 160+c(0,1,2,3,4,5,6,7,8,9,10)*10
view <- c(1930,2010, 160,250); orig <- c(1930,160)
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
lines(hm$V1,hm$V2); lines(hf$V1,hf$V2)
points(hm$V1,hm$V2, pch=".", cex=4, col="blue")
points(hf$V1,hf$V2, pch=".", cex=4, col="red")
 # Per rappresentare i numeri indici ne metto i valori in indm e
indf e poi procedo nel solito modo, cambiando scala e tacche:
indm <- hm$V2/hm$V2[1]*100; indf <- hf$V2/hf$V2[1]*100
ty <- 100+c(0,5,10,15,20,25,30)
view <- c(1930,2010, 100,130); orig <- c(1930,100)
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
lines(hm$V1,indm); lines(hf$V1,indf)
points(hm$V1,indm, pch=".", cex=4, col="blue")
points(hf$V1,indf, pch=".", cex=4, col="red")
# Per rappresentare i numeri indici ne metto i valori in indm e
indf e poi procedo nel solito modo, cambiando scala e tacche:
indm <- hm$V2/hm$V2[1]*100; indf <- hf$V2/hf$V2[1]*100
ty <- 100+c(0,5,10,15,20,25,30)
view <- c(1930,2010, 100,130); orig <- c(1930,100)
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
lines(hm$V1,indm); lines(hf$V1,indf)
points(hm$V1,indm, pch=".", cex=4, col="blue")
points(hf$V1,indf, pch=".", cex=4, col="red")
 #
# >> ALTRE CURVE <<
# Vediamo, prima, come fare il grafico di più funzioni (vedi LA-2-q.36):
# basta aggiungere più comandi plot.
F <- function(x) x*1.25; G <- function(x) x*0.8
view <- c(-25,160, -25,160); orig <- c(0,0)
tx <- c(-2,-1,0,1,2,3,4,5,6,7,8,9,10,11,12,13,14,15,16)*10; ty <- tx
# i soliti comandi:
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
# quindi:
curve(F, add=TRUE,col="blue"); curve(G, add=TRUE,col="red")
H <- function(x) F(G(x)); K <- function(x) G(F(x))
curve(H, add=TRUE,col="green"); curve(K, add=TRUE,col="magenta")
#
# >> ALTRE CURVE <<
# Vediamo, prima, come fare il grafico di più funzioni (vedi LA-2-q.36):
# basta aggiungere più comandi plot.
F <- function(x) x*1.25; G <- function(x) x*0.8
view <- c(-25,160, -25,160); orig <- c(0,0)
tx <- c(-2,-1,0,1,2,3,4,5,6,7,8,9,10,11,12,13,14,15,16)*10; ty <- tx
# i soliti comandi:
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
# quindi:
curve(F, add=TRUE,col="blue"); curve(G, add=TRUE,col="red")
H <- function(x) F(G(x)); K <- function(x) G(F(x))
curve(H, add=TRUE,col="green"); curve(K, add=TRUE,col="magenta")
 #
# Per fare il grafico di una curva che non sia una funzione (vedi LA-2-e8)
# posso usare il comando lines. Ecco il grafico della relazione
# inversa di una funzione (spezzo il grafico in 1000 segmentini)
view <- c(-4,10, -4,10); orig <- c(0,0)
tx <- c(-4,-3,-2,-1,0,1,2,3,4,5,6,7,8,9,10); ty <- tx
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="",asp=1)
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
f <- function(x) x^2; curve(f,add=TRUE,col="blue")
t1<-view[1]; t2<-view[2]; punti<-1000; t<- seq(t1,t2,(t2-t1)/punti)
lines(f(t),t,col="green4")
#
# Per fare il grafico di una curva che non sia una funzione (vedi LA-2-e8)
# posso usare il comando lines. Ecco il grafico della relazione
# inversa di una funzione (spezzo il grafico in 1000 segmentini)
view <- c(-4,10, -4,10); orig <- c(0,0)
tx <- c(-4,-3,-2,-1,0,1,2,3,4,5,6,7,8,9,10); ty <- tx
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="",asp=1)
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
f <- function(x) x^2; curve(f,add=TRUE,col="blue")
t1<-view[1]; t2<-view[2]; punti<-1000; t<- seq(t1,t2,(t2-t1)/punti)
lines(f(t),t,col="green4")
 #
# >> PRIME STATISTICHE <<
# Elaborazioni riferite a LS-3-e9,…; è evidente
# che cosa fanno i vari comandi.
alu <- c(156,168,162,150,167,157,170,157,159,164,157,165,
163,165,166,160,163,162,155)
length(alu);sum(alu); min(alu);max(alu); range(alu);sort(alu); mean(alu);median(alu)
[1] 19
[1] 3066
[1] 150
[1] 170
[1] 150 170
[1] 150 155 156 157 157 157 159 160 162 162 163 163 164 165 165 166 167
[18] 168 170
[1] 161.3684
[1] 162
# Il comando che segue rappresenta automaticamente i dati su una specie
# di istogramma, riportando anche i valori dei dati ("stem" è il gambo,
# su cui poggiano le foglie)
stem(alu)
The decimal point is 1 digit(s) to the right of the |
15 | 0
15 | 567779
16 | 022334
16 | 55678
17 | 0
# summary riporta min e max, media e mediana, 1° e 3° quartile, ossia
# il 25° e il 75° percentile
summary(alu)
Min. 1st Qu. Median Mean 3rd Qu. Max.
150.0 157.0 162.0 161.4 165.0 170.0
# A differenza di barplot, hist traccia l'istogramma di dati che, qui,
# vengono classificati mediante seq (che abbiamo già usato): seq(A,B,h)
# genera la sequenza di dati che va, di h in h, da A a B; l'opzione
# right=FALSE specifica che i sottintervalli sono del tipo [.,.), ossia
# aperti a destra (e chiusi a sinistra)
# boxplot traccia il box-plot (con l'opzione scelta, orizzontale)
hist(alu,seq(150,171,3),right=FALSE,col="yellow",xlab="",ylab="",main="")
boxplot(alu, horizontal=TRUE)
#
# >> PRIME STATISTICHE <<
# Elaborazioni riferite a LS-3-e9,…; è evidente
# che cosa fanno i vari comandi.
alu <- c(156,168,162,150,167,157,170,157,159,164,157,165,
163,165,166,160,163,162,155)
length(alu);sum(alu); min(alu);max(alu); range(alu);sort(alu); mean(alu);median(alu)
[1] 19
[1] 3066
[1] 150
[1] 170
[1] 150 170
[1] 150 155 156 157 157 157 159 160 162 162 163 163 164 165 165 166 167
[18] 168 170
[1] 161.3684
[1] 162
# Il comando che segue rappresenta automaticamente i dati su una specie
# di istogramma, riportando anche i valori dei dati ("stem" è il gambo,
# su cui poggiano le foglie)
stem(alu)
The decimal point is 1 digit(s) to the right of the |
15 | 0
15 | 567779
16 | 022334
16 | 55678
17 | 0
# summary riporta min e max, media e mediana, 1° e 3° quartile, ossia
# il 25° e il 75° percentile
summary(alu)
Min. 1st Qu. Median Mean 3rd Qu. Max.
150.0 157.0 162.0 161.4 165.0 170.0
# A differenza di barplot, hist traccia l'istogramma di dati che, qui,
# vengono classificati mediante seq (che abbiamo già usato): seq(A,B,h)
# genera la sequenza di dati che va, di h in h, da A a B; l'opzione
# right=FALSE specifica che i sottintervalli sono del tipo [.,.), ossia
# aperti a destra (e chiusi a sinistra)
# boxplot traccia il box-plot (con l'opzione scelta, orizzontale)
hist(alu,seq(150,171,3),right=FALSE,col="yellow",xlab="",ylab="",main="")
boxplot(alu, horizontal=TRUE)
 # Vediamo come confrontare più boxplot
alu2 <- c(172,162,150,167,157,170,157,159,164,157,165,163,165,
166,160,163,162,155)
alu3 <- c(172,162,160,167,157,170,157,159,164,157,165,163,165,
166,160,163,162,180)
boxplot(alu, alu2, alu3)
# a destra la figura ottenuta con:
boxplot(alu, alu2, alu3, col="grey80",show.names=FALSE)
# Vediamo come confrontare più boxplot
alu2 <- c(172,162,150,167,157,170,157,159,164,157,165,163,165,
166,160,163,162,155)
alu3 <- c(172,162,160,167,157,170,157,159,164,157,165,163,165,
166,160,163,162,180)
boxplot(alu, alu2, alu3)
# a destra la figura ottenuta con:
boxplot(alu, alu2, alu3, col="grey80",show.names=FALSE)
 # Volendo possiamo anche sovrapporre più istogrammi, differenziandone
# l'aspetto con vari comandi; ecco un esempio:
interv <- seq(150,183,3); tacchey <- seq(0,6,1)
hist(alu,interv,right=FALSE,col="yellow",xlab="",ylab="",main="",axes=FALSE)
axis(1,pos=0,col="blue",label=TRUE, at=interv)
axis(2,pos=150,col="blue",label=TRUE, at=tacchey)
hist(alu2,interv,right=FALSE,xlab="",ylab="",main="",border="blue",add=TRUE,angle=135,density=14)
hist(alu3,interv,right=FALSE,xlab="",ylab="",main="",border="red",add=TRUE,angle=45,density=7)
# add=TRUE consente di aggiungere un istogramma alla figura già tracciata;
# angle e density fanno tracciare segmenti sulle colonne con varia
# inclinazione (in gradi) e varia densità (segmenti per pollice)
# axes=FALSE mi consente di non tracciare gli assi automaticamente;
# li ho tracciati col comando axis
# Volendo possiamo anche sovrapporre più istogrammi, differenziandone
# l'aspetto con vari comandi; ecco un esempio:
interv <- seq(150,183,3); tacchey <- seq(0,6,1)
hist(alu,interv,right=FALSE,col="yellow",xlab="",ylab="",main="",axes=FALSE)
axis(1,pos=0,col="blue",label=TRUE, at=interv)
axis(2,pos=150,col="blue",label=TRUE, at=tacchey)
hist(alu2,interv,right=FALSE,xlab="",ylab="",main="",border="blue",add=TRUE,angle=135,density=14)
hist(alu3,interv,right=FALSE,xlab="",ylab="",main="",border="red",add=TRUE,angle=45,density=7)
# add=TRUE consente di aggiungere un istogramma alla figura già tracciata;
# angle e density fanno tracciare segmenti sulle colonne con varia
# inclinazione (in gradi) e varia densità (segmenti per pollice)
# axes=FALSE mi consente di non tracciare gli assi automaticamente;
# li ho tracciati col comando axis
 # L'esercizio LS-3-e11 fa riferimento ad un file; prima di caricarlo conviene
# esaminarlo; col comando readLines possiamo leggerne un po' di righe,
# ad es. 5, per vedere come è fatto
readLines("http://macosa.dima.unige.it/om/prg/stf/t-sec.stf", n=5)
[1] "'commento: misure delle durata di 1 sec cronometrate manualmente"
[2] "111"
[3] "103"
[4] "109"
[5] "97"
# Con scan posso, volendo, esaminarlo (in questo caso i dati sono
# pochi e possiamo farlo), e posso caricarlo in una variabile, ad es.
# DATI; in ogni caso salto (con skip=1) la prima riga, di commento
DATI <- scan("http://macosa.dima.unige.it/om/prg/stf/t-sec.stf", skip=1)
length(DATI)
[1] 47
DATI
[1] 111 103 109 97 99 110 99 103 109 106 109 112 98 110
[15] 90 98 96 90 96 90 109 103 96 97 96 96 109 103
[29] 98 94 115 78 85 96 90 121 98 103 97 90 98 129
[43] 96 68 91 80 102
# I dati era tempi in secondi, troncati agli interi. Per avere
# rappresentazioni corrette possiamo aggiungere 1/2 ad essi:
DATI <- DATI+1/2; DATI
[1] 111.5 103.5 109.5 97.5 99.5 110.5 99.5 103.5 109.5
[10] 106.5 109.5 112.5 98.5 110.5 90.5 98.5 96.5 90.5
[19] 96.5 90.5 109.5 103.5 96.5 97.5 96.5 96.5 109.5
[28] 103.5 98.5 94.5 115.5 78.5 85.5 96.5 90.5 121.5
[37] 98.5 103.5 97.5 90.5 98.5 129.5 96.5 68.5 91.5
[46] 80.5 102.5
min(DATI); max(DATI); mean(DATI); median(DATI)
[1] 68.5
[1] 129.5
[1] 99.92553
[1] 98.5
# l'istogramma, in classi ampie 10, e il box-plot, in giallo.
hist(DATI,seq(65,135,10),right=FALSE,col="yellow",xlab="",main="")
abline(h=c(5,10,15,20,25),col="red",lty=3)
# la griglia è stata aggiunta con i soliti comandi.
boxplot(DATI, horizontal=TRUE, col="yellow")
# L'esercizio LS-3-e11 fa riferimento ad un file; prima di caricarlo conviene
# esaminarlo; col comando readLines possiamo leggerne un po' di righe,
# ad es. 5, per vedere come è fatto
readLines("http://macosa.dima.unige.it/om/prg/stf/t-sec.stf", n=5)
[1] "'commento: misure delle durata di 1 sec cronometrate manualmente"
[2] "111"
[3] "103"
[4] "109"
[5] "97"
# Con scan posso, volendo, esaminarlo (in questo caso i dati sono
# pochi e possiamo farlo), e posso caricarlo in una variabile, ad es.
# DATI; in ogni caso salto (con skip=1) la prima riga, di commento
DATI <- scan("http://macosa.dima.unige.it/om/prg/stf/t-sec.stf", skip=1)
length(DATI)
[1] 47
DATI
[1] 111 103 109 97 99 110 99 103 109 106 109 112 98 110
[15] 90 98 96 90 96 90 109 103 96 97 96 96 109 103
[29] 98 94 115 78 85 96 90 121 98 103 97 90 98 129
[43] 96 68 91 80 102
# I dati era tempi in secondi, troncati agli interi. Per avere
# rappresentazioni corrette possiamo aggiungere 1/2 ad essi:
DATI <- DATI+1/2; DATI
[1] 111.5 103.5 109.5 97.5 99.5 110.5 99.5 103.5 109.5
[10] 106.5 109.5 112.5 98.5 110.5 90.5 98.5 96.5 90.5
[19] 96.5 90.5 109.5 103.5 96.5 97.5 96.5 96.5 109.5
[28] 103.5 98.5 94.5 115.5 78.5 85.5 96.5 90.5 121.5
[37] 98.5 103.5 97.5 90.5 98.5 129.5 96.5 68.5 91.5
[46] 80.5 102.5
min(DATI); max(DATI); mean(DATI); median(DATI)
[1] 68.5
[1] 129.5
[1] 99.92553
[1] 98.5
# l'istogramma, in classi ampie 10, e il box-plot, in giallo.
hist(DATI,seq(65,135,10),right=FALSE,col="yellow",xlab="",main="")
abline(h=c(5,10,15,20,25),col="red",lty=3)
# la griglia è stata aggiunta con i soliti comandi.
boxplot(DATI, horizontal=TRUE, col="yellow")
 #
# Sotto, il box-plot che avrei ottenuto con Stat:
··········|------------|=|========|----|············
+-----------------------------+------------------------+
65 100 130
# la sintesi, e il 5° e il 95° percentile
summary(DATI); quantile(DATI,0.05); quantile(DATI,0.95)
Min. 1st Qu. Median Mean 3rd Qu. Max.
68.50 96.50 98.50 99.93 108.00 129.50
5% 82
95% 114.6
# NOTA: Stat traccia i boxplot segnando pure il 5º e il 95º percentile;
# R traccia solo il 25º e il 75º percentile evidenziando poi con dei
# pallini gli eventuali dati che precedono il 25º o seguono il 75º per
# più di 1.5 volte la distanza interquartile, dando in modo alternativo
# l'idea della eventuale dispersione dei dati. Volendo, con l'opzione
# RANGE=0 si elimina questa visualizzazione; vedi il secondo boxplot:
#
# Sotto, il box-plot che avrei ottenuto con Stat:
··········|------------|=|========|----|············
+-----------------------------+------------------------+
65 100 130
# la sintesi, e il 5° e il 95° percentile
summary(DATI); quantile(DATI,0.05); quantile(DATI,0.95)
Min. 1st Qu. Median Mean 3rd Qu. Max.
68.50 96.50 98.50 99.93 108.00 129.50
5% 82
95% 114.6
# NOTA: Stat traccia i boxplot segnando pure il 5º e il 95º percentile;
# R traccia solo il 25º e il 75º percentile evidenziando poi con dei
# pallini gli eventuali dati che precedono il 25º o seguono il 75º per
# più di 1.5 volte la distanza interquartile, dando in modo alternativo
# l'idea della eventuale dispersione dei dati. Volendo, con l'opzione
# RANGE=0 si elimina questa visualizzazione; vedi il secondo boxplot:
 # L'analisi con R dei dati di LS-3-e12:
readLines("http://macosa.dima.unige.it/om/prg/stf/t-arrivi.stf", n=5)
[1] "'commento: distanze temporali tra arrivi successivi a uno sportello"
[2] "15"
[3] "29"
[4] "4"
[5] "99"
DATI <- scan("http://macosa.dima.unige.it/om/prg/stf/t-arrivi.stf", skip=1)
length(DATI)
[1] 134
DATI <- DATI+1/2
min(DATI); max(DATI); mean(DATI); median(DATI)
[1] 1.5
[1] 173.5
[1] 29.55224
[1] 21.5
hist(DATI,seq(0,180,10),right=FALSE,col="yellow",xlab="",main="")
# L'analisi con R dei dati di LS-3-e12:
readLines("http://macosa.dima.unige.it/om/prg/stf/t-arrivi.stf", n=5)
[1] "'commento: distanze temporali tra arrivi successivi a uno sportello"
[2] "15"
[3] "29"
[4] "4"
[5] "99"
DATI <- scan("http://macosa.dima.unige.it/om/prg/stf/t-arrivi.stf", skip=1)
length(DATI)
[1] 134
DATI <- DATI+1/2
min(DATI); max(DATI); mean(DATI); median(DATI)
[1] 1.5
[1] 173.5
[1] 29.55224
[1] 21.5
hist(DATI,seq(0,180,10),right=FALSE,col="yellow",xlab="",main="")
 # per avere l'istogramma delle densità di frequenza:
> hist(DATI,seq(0,180,10),right=FALSE,col="yellow",xlab="",main="",probability=TRUE)
# che poi rappresento scegliendo la scala verticale
hist(DATI,seq(0,180,10),ylim=c(0,0.03),right=FALSE,col="yellow",xlab="",main="",probability=TRUE)
# per avere l'istogramma delle densità di frequenza:
> hist(DATI,seq(0,180,10),right=FALSE,col="yellow",xlab="",main="",probability=TRUE)
# che poi rappresento scegliendo la scala verticale
hist(DATI,seq(0,180,10),ylim=c(0,0.03),right=FALSE,col="yellow",xlab="",main="",probability=TRUE)
 # Sull'istogramma è sovrapposto il grafico dell'esponenziale negativa
# (discussa nella guida alla scheda) e la posizione di media e mediana, cose
# ottenute coi comandi seguenti:
w <- 1/mean(DATI); f <- function(x) w*exp(-w*x)
plot(f,0,180,add=TRUE)
abline(v=mean(DATI),col="red"); abline(v=median(DATI),col="blue")
#
# Ecco come analizzare con R i dati di LS-3-e13, già classificati in
# intervalli
# Metto in una variabile (freq) le frequenze, in un'altra (num_int)
# il numero di intervalli, in una terza (interv) gli estremi degli
# intervallo, e poi metto la riga successiva (le righe in viola le
# copio e incollo, senza preoccuparmi di capirne i dettagli)
morti <- c(46,25,58,186,870,1071,3124)
interv <- c(0,1,15,25,45,65,75,100) # un dato in piu' di freq
totale <- sum(morti); fr_perc <- morti/totale*100; fr_perc
[1] 0.8550186 0.4646840 1.0780669 3.4572491 16.1710037 19.9070632
[7] 58.0669145
# Arrotondo: metto digits=2 in quanto il dato più piccolo 0.464... lo
# voglio arrotondare a 0.46, ossia a 2 cifre
print(fr_perc,digits=2)
[1] 0.86 0.46 1.08 3.46 16.17 19.91 58.07
# porto i dati a 500 mila per migliorare l'elaborazione
rap <- 1e5/sum(morti); freq <- morti*rap; num_int <- length(freq)
dati <- c(seq(interv[1],interv[2],by=(interv[2]-interv[1])/freq[1]))
for(i in 2:num_int){dati<- c(dati,seq(interv[i],interv[i+1],by=(interv[i+1]-interv[i])/freq[i]))}
hist(dati,interv,right=FALSE,xlab="",main="")
# Sull'istogramma è sovrapposto il grafico dell'esponenziale negativa
# (discussa nella guida alla scheda) e la posizione di media e mediana, cose
# ottenute coi comandi seguenti:
w <- 1/mean(DATI); f <- function(x) w*exp(-w*x)
plot(f,0,180,add=TRUE)
abline(v=mean(DATI),col="red"); abline(v=median(DATI),col="blue")
#
# Ecco come analizzare con R i dati di LS-3-e13, già classificati in
# intervalli
# Metto in una variabile (freq) le frequenze, in un'altra (num_int)
# il numero di intervalli, in una terza (interv) gli estremi degli
# intervallo, e poi metto la riga successiva (le righe in viola le
# copio e incollo, senza preoccuparmi di capirne i dettagli)
morti <- c(46,25,58,186,870,1071,3124)
interv <- c(0,1,15,25,45,65,75,100) # un dato in piu' di freq
totale <- sum(morti); fr_perc <- morti/totale*100; fr_perc
[1] 0.8550186 0.4646840 1.0780669 3.4572491 16.1710037 19.9070632
[7] 58.0669145
# Arrotondo: metto digits=2 in quanto il dato più piccolo 0.464... lo
# voglio arrotondare a 0.46, ossia a 2 cifre
print(fr_perc,digits=2)
[1] 0.86 0.46 1.08 3.46 16.17 19.91 58.07
# porto i dati a 500 mila per migliorare l'elaborazione
rap <- 1e5/sum(morti); freq <- morti*rap; num_int <- length(freq)
dati <- c(seq(interv[1],interv[2],by=(interv[2]-interv[1])/freq[1]))
for(i in 2:num_int){dati<- c(dati,seq(interv[i],interv[i+1],by=(interv[i+1]-interv[i])/freq[i]))}
hist(dati,interv,right=FALSE,xlab="",main="")
 # In realtà l'istogramma è stato colorato:
hist(dati,interv,right=FALSE,col="yellow",xlab="",main="")
# Ecco come aggiungere una "griglia"
abline(v=interv,h=seq(0.005,0.03,0.005),lty=3,col="grey50")
# In realtà l'istogramma è stato colorato:
hist(dati,interv,right=FALSE,col="yellow",xlab="",main="")
# Ecco come aggiungere una "griglia"
abline(v=interv,h=seq(0.005,0.03,0.005),lty=3,col="grey50")
 mean(dati); median(dati)
# Alternativa:
tacchey <- c(0,0.005,0.01,0.015,0.02,0.025)
hist(dati,interv,col="yellow",axes=FALSE,xlab="",main="",ylim=c(0,max(tacchey)))
abline(h = tacchey, col="grey60",lty=3)
axis(1,pos=0,col="blue",label=TRUE, at=interv)
axis(2,pos=interv[1],col="blue",label=TRUE, at=tacchey)
mean(dati); median(dati)
# Alternativa:
tacchey <- c(0,0.005,0.01,0.015,0.02,0.025)
hist(dati,interv,col="yellow",axes=FALSE,xlab="",main="",ylim=c(0,max(tacchey)))
abline(h = tacchey, col="grey60",lty=3)
axis(1,pos=0,col="blue",label=TRUE, at=interv)
axis(2,pos=interv[1],col="blue",label=TRUE, at=tacchey)
 # modifico i dati (e copio le altre righe); ottengo:
morti <- c(58,186,870,1071,3124); interv <- c(15,25,45,65,75,100)
...
# modifico i dati (e copio le altre righe); ottengo:
morti <- c(58,186,870,1071,3124); interv <- c(15,25,45,65,75,100)
...
 #
# altezze dei ventenni italiani nel 1976, discusse in vedi LS-3-§4
# analisi dei dati, svolta come sopra
num_int <- 9; freq <- c(7,29,112,240,289,204,89,25,5)
interv <- c(150,155,160,165,170,175,180,185,190,200)
totale <- sum(freq); fr_perc <- freq/totale*100; fr_perc
[1] 0.7 2.9 11.2 24.0 28.9 20.4 8.9 2.5 0.5
dati <- c(seq(interv[1],interv[2],by=(interv[2]-interv[1])/freq[1]))
for (i in 2:num_int) {dati <- c(dati,seq(interv[i],interv[i+1],by=(interv[i+1]-interv[i])/freq[i]))}
hist(dati,interv)
# ottenuto l'istogramma sotto a sinistra, posso scegliere la
# scala verticale e proseguire ottenedo l'istogramma destro
#
# altezze dei ventenni italiani nel 1976, discusse in vedi LS-3-§4
# analisi dei dati, svolta come sopra
num_int <- 9; freq <- c(7,29,112,240,289,204,89,25,5)
interv <- c(150,155,160,165,170,175,180,185,190,200)
totale <- sum(freq); fr_perc <- freq/totale*100; fr_perc
[1] 0.7 2.9 11.2 24.0 28.9 20.4 8.9 2.5 0.5
dati <- c(seq(interv[1],interv[2],by=(interv[2]-interv[1])/freq[1]))
for (i in 2:num_int) {dati <- c(dati,seq(interv[i],interv[i+1],by=(interv[i+1]-interv[i])/freq[i]))}
hist(dati,interv)
# ottenuto l'istogramma sotto a sinistra, posso scegliere la
# scala verticale e proseguire ottenedo l'istogramma destro
 tacchey <- c(0,0.5,1,1.5,2,2.5,3,3.5,4,4.5,5,5.5,6)/100
hist(dati,interv,col="yellow",axes=FALSE,xlab="",main="",ylim=c(0,max(tacchey)))
abline(h = tacchey, col="grey60",lty=3)
axis(1,pos=0,col="blue",label=TRUE, at=interv)
axis(2,pos=interv[1],col="blue",label=TRUE, at=tacchey)
# altre valutazioni statistiche dei dati
summary(dati)
Min. 1st Qu. Median Mean 3rd Qu. Max.
150.0 167.1 171.9 172.0 176.8 200.0
media: 172.0025 mediana = 171.9377163
5% :160.625 25% :167.125 50% :171.9377163
95% :183.8764045 75% :176.7892157
···········|------|====|====|------|·················
tacchey <- c(0,0.5,1,1.5,2,2.5,3,3.5,4,4.5,5,5.5,6)/100
hist(dati,interv,col="yellow",axes=FALSE,xlab="",main="",ylim=c(0,max(tacchey)))
abline(h = tacchey, col="grey60",lty=3)
axis(1,pos=0,col="blue",label=TRUE, at=interv)
axis(2,pos=interv[1],col="blue",label=TRUE, at=tacchey)
# altre valutazioni statistiche dei dati
summary(dati)
Min. 1st Qu. Median Mean 3rd Qu. Max.
150.0 167.1 171.9 172.0 176.8 200.0
media: 172.0025 mediana = 171.9377163
5% :160.625 25% :167.125 50% :171.9377163
95% :183.8764045 75% :176.7892157
···········|------|====|====|------|·················
 #
# >> CALCOLI VARI <<
# Struttura di un termine (vedi LA-3-§2)
G <- function(x) (2^3*-x)/(9-1)+x*5; G(3); G(-5)
[1] 12
[1] -20
x <- 3; v0<-2^3; v1<- -x; v2<-v0*v1; v3<-9-1; v4<-v2/v3; v5<-x*5; v6<-v4+v5; v6
[1] 12
x <- -5; v0<-2^3; v1<- -x; v2<-v0*v1; v3<-9-1; v4<-v2/v3; v5<-x*5; v6<-v4+v5; v6
[1] -20
# Divisori e scomposizione in numeri primi (vedi LA-3-§4); floor(x) dà
# il massimo intero minore o uguale ad x; in una condizione, come quella
# che segue un if, occorre usare == invece di =.
n <- 1580; for(k in 1:n) if (n/k==floor(n/k)) print(k)
[1] 1
[1] 2
[1] 4
[1] 5
[1] 10
[1] 20
[1] 79
[1] 158
[1] 316
[1] 395
[1] 790
[1] 1580
# Occorre usare { e }quando si vuole delimitare l'esecuzione ad alcuni
# comandi; nell'esempio che segue servono per far eseguire print(k)
# e n<-n/k solo se la condizione è verificata
# while significa "fintanto che"
n<-1580; k<-2; while (k<=n) if (n/k==floor(n/k)) {print(k); n<-n/k} else k<-k+1
[1] 2
[1] 2
[1] 5
[1] 79
# La distanza dall'origine (vedi LA-3-e8) è definibile:
D <- function(x,y) sqrt(x^2+y^2)
D(1,2)
[1] 2.236068
# viene visualizzata un'approssimazione del risultato; il programma
# ne elabora in realtà una più precisa (2.236067977...), come si
# vede trasformando l'uscita in forma intera:
for (i in 8:12) print(D(1,2)*10^i)
[1] 223606798
[1] 2236067977
[1] 22360679775
[1] 223606797750
[1] 2.236068e+12
# In R le assegnazioni (vedi AL-§2) sono fatte usando <- o ->;
# occorre sempre esplicitare il simbolo di moltiplicazione, usando *
2·2
Error: unexpected input in "2·"
> 2*2
[1] 4
# Come già visto, chiamando la libreria MASS è possibile approssimare
# i numeri decimali mediante frazioni
library(MASS)
fractions(0.140140140140140140140); fractions(10.45454545454545454545)
[1] 140/999
[1] 115/11
# per passare da una forma frazionaria generata da fractions ad
# un numero usuale si può usare as.numeric:
x <- fractions(10.45454545454545454545); x
[1] 115/11
y <- as.numeric(x); y
[1] 10.45455
# ricordo che vengono visulizzate solo alcune cifre di quelle memorizzate:
y*1e10
[1] 104545454545
# Ecco come si potrebbero analizzare i dati in LA-4-§2 elaborati con un
# foglio di calcolo (array è una tabella; dim(3,1) specifica che
# è a 3 righe ed 1 colonna; data una tabella T l'elemento nella
# riga M e nella colonna N è indicato T[M,N])
iscr_97 <- array(c(181,146,245),dim=c(3,1))
iscr_98 <- array(c(154,126,140),dim=c(3,1))
ripe_97 <- array(c(81,38,48),dim=c(3,1))
ripe_98 <- array(c(36,35,37),dim=c(3,1))
totale <- array(c(iscr_97,rip_97,iscr_98,rip_98),dim=c(3,4)); totale
[,1] [,2] [,3] [,4]
[1,] 181 81 154 36
[2,] 146 38 126 35
[3,] 245 48 140 37
abband_1_2 <- totale[1,1]-totale[1,4]-totale[2,3]+totale[2,4]; abband_1_2
[1] 54
abband_2_3 <- totale[2,1]-totale[2,4]-totale[3,3]+totale[3,4]; abband_2_3
[1] 8
# Se voglio somme di righe o colonne posso precedere così:
sum(ripe_97)
[1] 167
sum(totale[1,])
[1] 452
sum(totale[,2])
[1] 167
# Calcoli con intervalli di indeterminazione (vedi IN-2-§4)
# moltiplicazione
xA <- 3.8; xB <- 3.9; yA <- 6.4; yB <- 6.5
p <- c(xA*yA, xA*yB, xB*yA, xB*yB); min(p); max(p)
[1] 24.32
[1] 25.35
# divisione
xA <- 1000; xB <- 1000; yA <- 920; yB <- 940
d <- c(xA/yA, xA/yB, xB/yA, xB/yB); min(d); max(d)
[1] 1.063830
[1] 1.086957
# calcolo dell'ipotenusa di un triangolo rettangolo (vedi PS-1-q.17)
c1A <- 53; c1B <- 54; c2A <- 30; c2B <- 31
ip1 <- sqrt(c1A^2+c2A^2); ip2 <- sqrt(c1B^2+c2B^2); ip1; ip2
[1] 60.90156
[1] 62.26556
# significato degli operatori logici (vedi PS-1-e4)
x<- 2; y<- 2; z<- 2; w<- 2; P<- x==y; Q<- z==w; c(!P, P&Q, P|Q)
[1] FALSE TRUE TRUE
x<- 2; y<- 2; z<- 2; w<- 1; P<- x==y; Q<- z==w; c(!P, P&Q, P|Q)
[1] FALSE FALSE TRUE
#
# >> FIGURE VARIE <<
# Alcune figure considerate in LA-4-§5
# Ecco come tracciare cerchi e quadrati vari usando il
# comando symbols
circ <- function(x,y,r,colore) symbols(x,y,circles=r,add=TRUE,fg=colore,inches = FALSE)
squa <- function(x,y,r,colore) symbols(x,y,squares=r,add=TRUE,fg=colore,inches = FALSE)
view <- c(-3,3, -3,3); orig <- c(0,0)
tx <- c(-3,-2,-1,0,1,2,3); ty <- tx
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="", asp=1)
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
circ(0,0,1,"blue"); circ(0,0,1.5,"red"); circ(-1,1,1.5,"magenta")
squa(0,0,1*2,"blue"); squa(0,0,1.5*2,"red"); squa(-1,1,1.5*2,"magenta")
#
# >> CALCOLI VARI <<
# Struttura di un termine (vedi LA-3-§2)
G <- function(x) (2^3*-x)/(9-1)+x*5; G(3); G(-5)
[1] 12
[1] -20
x <- 3; v0<-2^3; v1<- -x; v2<-v0*v1; v3<-9-1; v4<-v2/v3; v5<-x*5; v6<-v4+v5; v6
[1] 12
x <- -5; v0<-2^3; v1<- -x; v2<-v0*v1; v3<-9-1; v4<-v2/v3; v5<-x*5; v6<-v4+v5; v6
[1] -20
# Divisori e scomposizione in numeri primi (vedi LA-3-§4); floor(x) dà
# il massimo intero minore o uguale ad x; in una condizione, come quella
# che segue un if, occorre usare == invece di =.
n <- 1580; for(k in 1:n) if (n/k==floor(n/k)) print(k)
[1] 1
[1] 2
[1] 4
[1] 5
[1] 10
[1] 20
[1] 79
[1] 158
[1] 316
[1] 395
[1] 790
[1] 1580
# Occorre usare { e }quando si vuole delimitare l'esecuzione ad alcuni
# comandi; nell'esempio che segue servono per far eseguire print(k)
# e n<-n/k solo se la condizione è verificata
# while significa "fintanto che"
n<-1580; k<-2; while (k<=n) if (n/k==floor(n/k)) {print(k); n<-n/k} else k<-k+1
[1] 2
[1] 2
[1] 5
[1] 79
# La distanza dall'origine (vedi LA-3-e8) è definibile:
D <- function(x,y) sqrt(x^2+y^2)
D(1,2)
[1] 2.236068
# viene visualizzata un'approssimazione del risultato; il programma
# ne elabora in realtà una più precisa (2.236067977...), come si
# vede trasformando l'uscita in forma intera:
for (i in 8:12) print(D(1,2)*10^i)
[1] 223606798
[1] 2236067977
[1] 22360679775
[1] 223606797750
[1] 2.236068e+12
# In R le assegnazioni (vedi AL-§2) sono fatte usando <- o ->;
# occorre sempre esplicitare il simbolo di moltiplicazione, usando *
2·2
Error: unexpected input in "2·"
> 2*2
[1] 4
# Come già visto, chiamando la libreria MASS è possibile approssimare
# i numeri decimali mediante frazioni
library(MASS)
fractions(0.140140140140140140140); fractions(10.45454545454545454545)
[1] 140/999
[1] 115/11
# per passare da una forma frazionaria generata da fractions ad
# un numero usuale si può usare as.numeric:
x <- fractions(10.45454545454545454545); x
[1] 115/11
y <- as.numeric(x); y
[1] 10.45455
# ricordo che vengono visulizzate solo alcune cifre di quelle memorizzate:
y*1e10
[1] 104545454545
# Ecco come si potrebbero analizzare i dati in LA-4-§2 elaborati con un
# foglio di calcolo (array è una tabella; dim(3,1) specifica che
# è a 3 righe ed 1 colonna; data una tabella T l'elemento nella
# riga M e nella colonna N è indicato T[M,N])
iscr_97 <- array(c(181,146,245),dim=c(3,1))
iscr_98 <- array(c(154,126,140),dim=c(3,1))
ripe_97 <- array(c(81,38,48),dim=c(3,1))
ripe_98 <- array(c(36,35,37),dim=c(3,1))
totale <- array(c(iscr_97,rip_97,iscr_98,rip_98),dim=c(3,4)); totale
[,1] [,2] [,3] [,4]
[1,] 181 81 154 36
[2,] 146 38 126 35
[3,] 245 48 140 37
abband_1_2 <- totale[1,1]-totale[1,4]-totale[2,3]+totale[2,4]; abband_1_2
[1] 54
abband_2_3 <- totale[2,1]-totale[2,4]-totale[3,3]+totale[3,4]; abband_2_3
[1] 8
# Se voglio somme di righe o colonne posso precedere così:
sum(ripe_97)
[1] 167
sum(totale[1,])
[1] 452
sum(totale[,2])
[1] 167
# Calcoli con intervalli di indeterminazione (vedi IN-2-§4)
# moltiplicazione
xA <- 3.8; xB <- 3.9; yA <- 6.4; yB <- 6.5
p <- c(xA*yA, xA*yB, xB*yA, xB*yB); min(p); max(p)
[1] 24.32
[1] 25.35
# divisione
xA <- 1000; xB <- 1000; yA <- 920; yB <- 940
d <- c(xA/yA, xA/yB, xB/yA, xB/yB); min(d); max(d)
[1] 1.063830
[1] 1.086957
# calcolo dell'ipotenusa di un triangolo rettangolo (vedi PS-1-q.17)
c1A <- 53; c1B <- 54; c2A <- 30; c2B <- 31
ip1 <- sqrt(c1A^2+c2A^2); ip2 <- sqrt(c1B^2+c2B^2); ip1; ip2
[1] 60.90156
[1] 62.26556
# significato degli operatori logici (vedi PS-1-e4)
x<- 2; y<- 2; z<- 2; w<- 2; P<- x==y; Q<- z==w; c(!P, P&Q, P|Q)
[1] FALSE TRUE TRUE
x<- 2; y<- 2; z<- 2; w<- 1; P<- x==y; Q<- z==w; c(!P, P&Q, P|Q)
[1] FALSE FALSE TRUE
#
# >> FIGURE VARIE <<
# Alcune figure considerate in LA-4-§5
# Ecco come tracciare cerchi e quadrati vari usando il
# comando symbols
circ <- function(x,y,r,colore) symbols(x,y,circles=r,add=TRUE,fg=colore,inches = FALSE)
squa <- function(x,y,r,colore) symbols(x,y,squares=r,add=TRUE,fg=colore,inches = FALSE)
view <- c(-3,3, -3,3); orig <- c(0,0)
tx <- c(-3,-2,-1,0,1,2,3); ty <- tx
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="", asp=1)
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
circ(0,0,1,"blue"); circ(0,0,1.5,"red"); circ(-1,1,1.5,"magenta")
squa(0,0,1*2,"blue"); squa(0,0,1.5*2,"red"); squa(-1,1,1.5*2,"magenta")
 # Ecco come calcolare le coordinate del cerchio di centro (0,0)
# e raggio 1 (vedrai più avanti il significato di cos e sin)
xc <- function(angolo) cos(angolo*pi/180)
yc <- function(angolo) sin(angolo*pi/180)
c(xc(30),yc(30)); c(xc(45),yc(45)); c(xc(0),yc(0)); c(xc(180),yc(180))
[1] 0.8660254 0.5000000
[1] 0.7071068 0.7071068
[1] 1 0
[1] -1.000000e+00 1.224606e-16 (è circa -1 0)
# ed ecco come tracciare tale cerchio in modo alternativo al precedente
view <- c(-3,3, -3,3); orig <- c(0,0)
tx <- c(-3,-2,-1,0,1,2,3); ty <- tx
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="", asp=1)
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
ang <- seq(0, 360, 360/1000)
lines(xc(ang), yc(ang))
# alcuni punti su di esso
points(xc(30),yc(30)); points(xc(-45),yc(-45)); points(xc(240),yc(240))
# alcune trasformazioni di esso
lines(xc(ang)+2,yc(ang)-1)
lines(xc(ang)/2+2,yc(ang)*2-1,col="red")
# ed un esagono regolare
ang1 <- seq(0, 360, 360/6); lines(xc(ang1)*2,yc(ang1)*2,col="blue")
# Ecco come calcolare le coordinate del cerchio di centro (0,0)
# e raggio 1 (vedrai più avanti il significato di cos e sin)
xc <- function(angolo) cos(angolo*pi/180)
yc <- function(angolo) sin(angolo*pi/180)
c(xc(30),yc(30)); c(xc(45),yc(45)); c(xc(0),yc(0)); c(xc(180),yc(180))
[1] 0.8660254 0.5000000
[1] 0.7071068 0.7071068
[1] 1 0
[1] -1.000000e+00 1.224606e-16 (è circa -1 0)
# ed ecco come tracciare tale cerchio in modo alternativo al precedente
view <- c(-3,3, -3,3); orig <- c(0,0)
tx <- c(-3,-2,-1,0,1,2,3); ty <- tx
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="", asp=1)
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
ang <- seq(0, 360, 360/1000)
lines(xc(ang), yc(ang))
# alcuni punti su di esso
points(xc(30),yc(30)); points(xc(-45),yc(-45)); points(xc(240),yc(240))
# alcune trasformazioni di esso
lines(xc(ang)+2,yc(ang)-1)
lines(xc(ang)/2+2,yc(ang)*2-1,col="red")
# ed un esagono regolare
ang1 <- seq(0, 360, 360/6); lines(xc(ang1)*2,yc(ang1)*2,col="blue")
 # La croce visualizzata è stata ottenuta (temporameante) cliccando
# sul punto indicato dopo aver scritto locator(1); in uscita si hanno
# le coordinate approssimative del punto cliccato.
locator(1)
$x
[1] -0.5211999
$y
[1] 0.5136463
# Volendo posso memorizzare le cordinate del punto cliccato aggiungendo
# $x e $y, come si vede qui sotto:
loc <- locator(1); xp <- loc$x; yp <- loc$y; xp; yp
[1] -0.5211999
[1] 0.5136463
# locator può essere usato anche per trovare graficamente soluzioni
# di equazioni; ecco ad es. come risolvere questo esercizio:
ty <- c(0,1,2,3,4,5,6,7,8,9,10)*10
tx <- c(0,1,2,3,4,5,6,7,8,9,10,11,12,13)
tx <- tx*10; k <- 100/124.2
f <- function(x) x*k
view <- c(0,130,0,100); orig <- c(0,0)
# i soliti comandi:
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
# il grafico di f e l'evidenziazione del punto (124.2, 100)
curve(f, add=TRUE, col="blue", from=0)
lines(c(124.2,124.2,0),c(0,100,100))
locator(1)
$x
[1] 34.41743
$y
[1] 27.96164
# La croce visualizzata è stata ottenuta (temporameante) cliccando
# sul punto indicato dopo aver scritto locator(1); in uscita si hanno
# le coordinate approssimative del punto cliccato.
locator(1)
$x
[1] -0.5211999
$y
[1] 0.5136463
# Volendo posso memorizzare le cordinate del punto cliccato aggiungendo
# $x e $y, come si vede qui sotto:
loc <- locator(1); xp <- loc$x; yp <- loc$y; xp; yp
[1] -0.5211999
[1] 0.5136463
# locator può essere usato anche per trovare graficamente soluzioni
# di equazioni; ecco ad es. come risolvere questo esercizio:
ty <- c(0,1,2,3,4,5,6,7,8,9,10)*10
tx <- c(0,1,2,3,4,5,6,7,8,9,10,11,12,13)
tx <- tx*10; k <- 100/124.2
f <- function(x) x*k
view <- c(0,130,0,100); orig <- c(0,0)
# i soliti comandi:
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
abline(h=ty,col="grey60",lty=3); abline(v=tx,col="grey60",lty=3)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
# il grafico di f e l'evidenziazione del punto (124.2, 100)
curve(f, add=TRUE, col="blue", from=0)
lines(c(124.2,124.2,0),c(0,100,100))
locator(1)
$x
[1] 34.41743
$y
[1] 27.96164
 # la figura a destra corrisponde al seguente "zoom" (con tacche
# più fitte):
view <- c(160,170,35,45)
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
curve(f, add=TRUE, col="blue", from=0)
ttx <- 160+tx/10
abline(v=ttx,col="orange",lty=3)
tty <- 35+ty/10
abline(h=tty,col="orange",lty=3)
locator(1)
$x
[1] 34.1082
$y
[1] 27.49286
# Concludo che al 2.75% corrispondono 3.4 miliardi. Ovviamente in questo
# caso è facile procedere numericamente: da x*k = 2.75 ho x = ...
2.75/k
[1] 3.4155
#
# Come ottenere altri poligoni regolari (vedi LA-4-e12) con comandi analoghi
# (xc e yc per avere le coordinate di un punto P del cerchio dato l'angolo
# in gradi di OP)
view <- c(-1,1, -1,1); orig <- c(0,0)
tx <- c(-1,-0.5,0,0.5,1); ty <- tx
abline(h = ty,col="grey"); abline(v = tx, col="grey")
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="", asp=1)
abline(h = ty,col="grey"); abline(v = tx, col="grey")
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
xc <- function(angolo) cos(angolo*pi/180); yc <- function(angolo) sin(angolo*pi/180)
ang <- function(n) seq(0, 360, 360/n)
for (n in 2:8) lines(xc(ang(n)),yc(ang(n)),col=n)
# la figura a destra corrisponde al seguente "zoom" (con tacche
# più fitte):
view <- c(160,170,35,45)
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
curve(f, add=TRUE, col="blue", from=0)
ttx <- 160+tx/10
abline(v=ttx,col="orange",lty=3)
tty <- 35+ty/10
abline(h=tty,col="orange",lty=3)
locator(1)
$x
[1] 34.1082
$y
[1] 27.49286
# Concludo che al 2.75% corrispondono 3.4 miliardi. Ovviamente in questo
# caso è facile procedere numericamente: da x*k = 2.75 ho x = ...
2.75/k
[1] 3.4155
#
# Come ottenere altri poligoni regolari (vedi LA-4-e12) con comandi analoghi
# (xc e yc per avere le coordinate di un punto P del cerchio dato l'angolo
# in gradi di OP)
view <- c(-1,1, -1,1); orig <- c(0,0)
tx <- c(-1,-0.5,0,0.5,1); ty <- tx
abline(h = ty,col="grey"); abline(v = tx, col="grey")
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="", asp=1)
abline(h = ty,col="grey"); abline(v = tx, col="grey")
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
xc <- function(angolo) cos(angolo*pi/180); yc <- function(angolo) sin(angolo*pi/180)
ang <- function(n) seq(0, 360, 360/n)
for (n in 2:8) lines(xc(ang(n)),yc(ang(n)),col=n)
 # un poligono qualunque si può tracciare col comando polygon (si
# mettono le x e le y in due collezioni di numeri:
view <- c(-3,3, -3,3); orig <- c(0,0)
tx <- c(-3,-2,-1,0,1,2,3); ty <- tx
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="", asp=1)
abline(h = ty,col="grey"); abline(v = tx, col="grey")
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
x <- c(0,1,2,2.5,-2); y <- c(1,3,-1.6,-2,-1)
polygon(x, y, col="yellow", border="blue")
# un poligono qualunque si può tracciare col comando polygon (si
# mettono le x e le y in due collezioni di numeri:
view <- c(-3,3, -3,3); orig <- c(0,0)
tx <- c(-3,-2,-1,0,1,2,3); ty <- tx
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="", asp=1)
abline(h = ty,col="grey"); abline(v = tx, col="grey")
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
x <- c(0,1,2,2.5,-2); y <- c(1,3,-1.6,-2,-1)
polygon(x, y, col="yellow", border="blue")
 #
# figure mediante caratteri (vedi PS-1-e8); text(x,y,"scritta") scrive
# la "scritta" centrata nel punto (x,y) della finestra grafica
view <- c(0,11, 0,11)
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="", asp=1)
for(i in 1:10) text(i,9,"O")
for(i in 9:5) text(1,i,"O")
for(i in 9:5) text(10,i,"O")
for(i in 1:10) text(i,4,"O")
#
# figure mediante caratteri (vedi PS-1-e8); text(x,y,"scritta") scrive
# la "scritta" centrata nel punto (x,y) della finestra grafica
view <- c(0,11, 0,11)
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="", asp=1)
for(i in 1:10) text(i,9,"O")
for(i in 9:5) text(1,i,"O")
for(i in 9:5) text(10,i,"O")
for(i in 1:10) text(i,4,"O")
 for(i in 10:5) text(1,i,"O")
for(i in 1:6) text(i,5,"O")
for(i in 1:6) text(i,11-i,"O")
#
# corona circolare (vedi MS-1-e7)
view <- c(-6,6, -6,6); orig <- c(0,0)
tx <- c(0,2,4,6,-2,-4,-6); ty <- tx
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="", asp=1)
abline(h = ty,col="grey"); abline(v = tx, col="grey")
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
fxy <- function(x,y) sqrt(x*x+y*y)
x1 <- -5; x2 <- 5; y1 <- -5; y2 <- 5
dx <- (x2-x1)/100; dy <- (y2-y1)/100; I <- 0:100; J <- 0:100
for(i in I) for(j in J) {x<-x1+dx*i;y<-y1+dy*j;if(fxy(x,y)<=5 & fxy(x,y)>=3) points(x,y,pch=".",col="blue")}
for(i in 10:5) text(1,i,"O")
for(i in 1:6) text(i,5,"O")
for(i in 1:6) text(i,11-i,"O")
#
# corona circolare (vedi MS-1-e7)
view <- c(-6,6, -6,6); orig <- c(0,0)
tx <- c(0,2,4,6,-2,-4,-6); ty <- tx
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="", asp=1)
abline(h = ty,col="grey"); abline(v = tx, col="grey")
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
fxy <- function(x,y) sqrt(x*x+y*y)
x1 <- -5; x2 <- 5; y1 <- -5; y2 <- 5
dx <- (x2-x1)/100; dy <- (y2-y1)/100; I <- 0:100; J <- 0:100
for(i in I) for(j in J) {x<-x1+dx*i;y<-y1+dy*j;if(fxy(x,y)<=5 & fxy(x,y)>=3) points(x,y,pch=".",col="blue")}
 #
# figure tridimensionali (vedi MS-1-e10)
# Ecco, a scatola nera, come realizzare il parallelepipedo dell'es.:
# in x ed y metto le coordinate di un rettangolo della faccia e in
# z metto una tabella 2*2 con le corrispondenti quote;
# nel comando persp indico gli intervalli per le x, le y e le z
# da rappresentare; ottengo la figura sotto a sinistra.
u<- c(3,3,3,3); z<- array(u,dim=c(2,2)); x<- c(6,8); y<- c(0,3)
PR <- persp(x,y,z,zlim=c(0,10),xlim=c(0,8),ylim=c(0,6),border="blue")
#
# figure tridimensionali (vedi MS-1-e10)
# Ecco, a scatola nera, come realizzare il parallelepipedo dell'es.:
# in x ed y metto le coordinate di un rettangolo della faccia e in
# z metto una tabella 2*2 con le corrispondenti quote;
# nel comando persp indico gli intervalli per le x, le y e le z
# da rappresentare; ottengo la figura sotto a sinistra.
u<- c(3,3,3,3); z<- array(u,dim=c(2,2)); x<- c(6,8); y<- c(0,3)
PR <- persp(x,y,z,zlim=c(0,10),xlim=c(0,8),ylim=c(0,6),border="blue")
 # poi aggiungo (nella stessa visione prospettica, che ho chiamato PR)
# col comando trans3d le coordinate delle altre facce
lines(trans3d(c(8,8,6,6,8),c(0,3,3,0,0),c(7,7,7,7,7),pmat=PR),col="red")
lines(trans3d(c(6,6),c(0,0),c(3,7),pmat=PR),col="green4")
lines(trans3d(c(6,6),c(3,3),c(3,7),pmat=PR),col="orange")
lines(trans3d(c(8,8),c(3,3),c(3,7),pmat=PR),col="brown")
#
# >> APPROFONDIMENTI <<
# Volendo, fra il "plot" e l'"abline" dei comandi in viola posso
# mettere comandi che mi infittiscono la griglia, ad es.
for (oriz in -3:25) abline(h=oriz,col="yellow")
for (vert in (-5:30)*1/5) abline(v=vert,col="yellow")
# poi aggiungo (nella stessa visione prospettica, che ho chiamato PR)
# col comando trans3d le coordinate delle altre facce
lines(trans3d(c(8,8,6,6,8),c(0,3,3,0,0),c(7,7,7,7,7),pmat=PR),col="red")
lines(trans3d(c(6,6),c(0,0),c(3,7),pmat=PR),col="green4")
lines(trans3d(c(6,6),c(3,3),c(3,7),pmat=PR),col="orange")
lines(trans3d(c(8,8),c(3,3),c(3,7),pmat=PR),col="brown")
#
# >> APPROFONDIMENTI <<
# Volendo, fra il "plot" e l'"abline" dei comandi in viola posso
# mettere comandi che mi infittiscono la griglia, ad es.
for (oriz in -3:25) abline(h=oriz,col="yellow")
for (vert in (-5:30)*1/5) abline(v=vert,col="yellow")
 # (-5:30)*1/5 sta per da -1 a 6 con passo 1/5; altro es.: per
# da 200 a 500 passo 50: 200/50=4, 500/50=10 quindi (4:10)*50
#
# Oppure mettere nel modo seguente gli estremi in cui tracciare
# le tacche e l'ampiezza di esse, e il colore:
ttx <- c(-3,25,1); tty <- c(-1,6,0.2); color="yellow"
# e quindi:
ttxx <- ttx/ttx[3]; ttyy <- tty/tty[3]
for (oriz in (ttxx[1]:ttxx[2])*ttx[3]) abline(h=oriz,col=color)
for (vert in (ttyy[1]:ttyy[2])*tty[3]) abline(v=vert,col=color)
#
# In definita posso mettere:
# descrizione delle funzioni
f <- function(x) 1.45*x; g <- function(x) 4.5*x
# rettangolo cartesiano e origine degli assi
view <- c(-1,6,-3,25); orig <- c(0,0)
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
# inizio, fine, ampiezza delle tacche
ttx <- c(-3,25, 1); tty <- c(-1,6, 0.2); color="yellow"
ttxx <- ttx/ttx[3]; ttyy <- tty/tty[3]
for (oriz in (ttxx[1]:ttxx[2])*ttx[3]) abline(h=oriz,col=color)
for (vert in (ttyy[1]:ttyy[2])*tty[3]) abline(v=vert,col=color)
ttx <- c(0,25, 5); tty <- c(-1,6, 1); color="grey"
ttxx <- ttx/ttx[3]; ttyy <- tty/tty[3]
for (oriz in (ttxx[1]:ttxx[2])*ttx[3]) abline(h=oriz,col=color)
for (vert in (ttyy[1]:ttyy[2])*tty[3]) abline(v=vert,col=color)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
# rappresentazione delle funzioni
curve(g, add=TRUE,col="green4", from=0, to=5)
curve(f, add=TRUE,col="blue", from=0, to=5)
# (-5:30)*1/5 sta per da -1 a 6 con passo 1/5; altro es.: per
# da 200 a 500 passo 50: 200/50=4, 500/50=10 quindi (4:10)*50
#
# Oppure mettere nel modo seguente gli estremi in cui tracciare
# le tacche e l'ampiezza di esse, e il colore:
ttx <- c(-3,25,1); tty <- c(-1,6,0.2); color="yellow"
# e quindi:
ttxx <- ttx/ttx[3]; ttyy <- tty/tty[3]
for (oriz in (ttxx[1]:ttxx[2])*ttx[3]) abline(h=oriz,col=color)
for (vert in (ttyy[1]:ttyy[2])*tty[3]) abline(v=vert,col=color)
#
# In definita posso mettere:
# descrizione delle funzioni
f <- function(x) 1.45*x; g <- function(x) 4.5*x
# rettangolo cartesiano e origine degli assi
view <- c(-1,6,-3,25); orig <- c(0,0)
plot(c(view[1],view[2]),c(view[3],view[4]),type="n",xlab="",ylab="")
# inizio, fine, ampiezza delle tacche
ttx <- c(-3,25, 1); tty <- c(-1,6, 0.2); color="yellow"
ttxx <- ttx/ttx[3]; ttyy <- tty/tty[3]
for (oriz in (ttxx[1]:ttxx[2])*ttx[3]) abline(h=oriz,col=color)
for (vert in (ttyy[1]:ttyy[2])*tty[3]) abline(v=vert,col=color)
ttx <- c(0,25, 5); tty <- c(-1,6, 1); color="grey"
ttxx <- ttx/ttx[3]; ttyy <- tty/tty[3]
for (oriz in (ttxx[1]:ttxx[2])*ttx[3]) abline(h=oriz,col=color)
for (vert in (ttyy[1]:ttyy[2])*tty[3]) abline(v=vert,col=color)
abline(h=orig[2],col="brown"); abline(v=orig[1],col="brown")
axis(1,pos=orig[2],label=FALSE,col="brown"); axis(2,pos=orig[1],label=FALSE,col="brown")
# rappresentazione delle funzioni
curve(g, add=TRUE,col="green4", from=0, to=5)
curve(f, add=TRUE,col="blue", from=0, to=5)